PCR en temps real
La PCR quantitativa, qPCR, Q-PCR[1] o PCR en temps real[2] és una variant de la reacció en cadena de la polimerasa (PCR) que es fa servir per amplificar i quantificar en termes absoluts el producte de l'amplificació, l'àcid desoxiribonucleic (ADN). Igual que en la PCR convencional, s'empra un motlle d'ADN, almenys un parell d'encebadors específics, dNTP, un amortidor de reacció adequat i una polimerasa d'ADN termoestable. S'afegeix a aquesta barreja una substància marcada amb un fluoròfor que, en un termociclador dotat de sensors per mesurar fluorescència, després de ser excitat a la longitud d'ona apropiada, permeti mesurar la taxa de generació d'un o més productes específics.[3] Aquest mesurament es fa després de cada cicle d'amplificació, motiu pel qual també se la denomina PCR en temps real. En molts casos el motlle emprat per la PCR quantitativa no és ADN des del principi, sinó que pot ser ADN complementari (ADNc) monocatenari, obtingut per retrotranscripció de l'àcid ribonucleic (ARN); en aquest cas, s'utilitzen les denominacions RT-PCR quantitativa, RT-PCR en temps real o RT-Q-PCR. No s'ha de confondre amb la tècnica de la PCR amb transcripció inversa[4] en què hi ha un pas de retrotranscripció d'ARN a ADN que no quantifica necessàriament el producte en temps real.[5]
Juntament amb els xips d'ADN, la PCR quantitativa és la metodologia més moderna per estudiar l'expressió gènica, tot i que altres mètodes tradicionals com el Northern blot també permeten observar-la amb una menor precisió.[6] La variabilitat introduïda a la quantificació, que comporta un marge d'error en l'estimació, deriva de la integritat de l'ADN, l'eficiència enzimàtica i molts altres factors, per la qual cosa s'han desenvolupat nombrosos sistemes d'estandardització. N'hi ha que quantifiquen de manera absoluta l'expressió gènica, però és més habitual que estiguin enfocats a la quantificació relativa del gen d'estudi respecte a un altre, denominat "normalitzador", que és seleccionat per la seva expressió gairebé constant. Aquests gens solen rebre el nom de "gens de manteniment" perquè sovint estant implicats en funcions bàsiques per a la supervivència cel·lular, cosa que sol comportar una expressió constitutiva.[7][8] Mesurant els gens d'interès a cada experiment i dividint el resultat per l'expressió del gen normalitzador seleccionat és possible comparar els gens objectiu, fins i tot sense conèixer-ne el nivell d'expressió en termes absoluts. Els gens normalitzadors més emprats són els que codifiquen proteïnes com la tubulina, la gliceraldehid-3-fosfat deshidrogenasa, l'albúmina, la ciclofilina o ARN ribosòmics.[6]
Fonaments[modifica]
La PCR quantitativa es duu a terme en un termociclador capaç de fer incidir sobre cada mostra un feix de llum d'una longitud d'ona determinada, així com de detectar la fluorescència emesa pel fluorocrom excitat. Aquest termociclador pot escalfar i refredar ràpidament les mostres, aprofitant les qualitats fisicoquímiques dels àcids nucleics i les qualitats enzimàtiques de l'ADN polimerasa.
El procés de la PCR consisteix en una sèrie de canvis de temperatura que repetits entre 25 i 40 vegades, anomenats cicles, cadascun amb un mínim de tres etapes: la primera, al voltant de 95 °C, permet la separació dels àcids nucleics de la doble cadena; la segona, a una temperatura al voltant de 50-60 °C, permet l'alineament dels encebadors a l'ADN motlle;[9] i la tercera, a 68-72 °C, facilita la polimerització per part de l'ADN polimerasa. A causa de la petitesa dels fragments amplificats generalment en aquest tipus de PCR, es pot ometre l'últim pas, car l'enzim és capaç d'amplificar durant la rampa entre la temperatura d'alineament i la de desnaturalització. A més, alguns termocicladors afegeixen a cada cicle uns segons a una altra temperatura, com ara 80 °C, per tal de reduir el soroll generat per la presència de dímers d'encebadors quan s'empra un colorant inespecífic. Les temperatures utilitzades i el temps aplicat a cada cicle depenen d'una gran varietat de paràmetres, com l'enzim utilitzat per la síntesi d'ADN, la concentració d'ions divalents i desoxiribonucleòtids (dNTP) en la reacció i la temperatura d'unió dels encebadors.[10]
Classificació[modifica]
Les tècniques de PCR quantitativa es poden classificar segons l'ús o bé de fluorocroms no específics o bé de sondes moleculars dependents de la seqüència.
En les tècniques basades en fluorocroms inespecífics es detecta la generació exponencial d'ADN bicatenari emprant un fluorocrom que s'hi uneix inespecíficament. Un exemple de colorant que permet aquesta detecció és el SYBR Green,[11] que quan és excitat amb llum blava (λmax = 488 nm) emet llum verda (λmax = 522 nm).[12] Presenta l'avantatge de requerir només un parell d'encebadors per dur a terme l'amplificació, cosa que abarateix el seu cost, però només és possible amplificar un producte a cada reacció. Les tècniques basades en sondes específiques utilitzen com a mínim un oligonucleòtid marcat amb fluorescència. Aquesta sonda està unida típicament a dos fluorocroms i hibrida a la zona intermèdia entre l'encebador directe (forward) i l'invers (reverse), és a dir, a l'amplicó. Així doncs, quan la sonda està intacta, presenten una transferència energètica de fluorescència per ressonància (FRET). Aquesta FRET no es produeix quan els dos fluorocroms estan distants a causa de la degradació de la sonda mitjançant l'activitat 5'- 3' exonucleasa de l'ADN polimerasa, o la separació física dels fluorocroms per un canvi en la conformació de la sonda. Això permet seguir el canvi del patró de fluorescència i deduir el nivell d'amplificació del gen.
Anàlisi de la temperatura de fusió[modifica]

Mitjançant l'ús un fluorocrom d'unió inespecífic a la doble cadena d'ADN, generalment SYBR Green, la Q-PCR permet identificar fragments amplificats d'ADN concrets a partir de la temperatura de fusió (també anomenat valor t, de l'anglès melting temperature), que és específica pel fragment amplificat que s'està buscant, i els resultats són obtinguts a partir de l'observació de la corba de dissociació de les mostres d'ADN analitzades.[14]
A diferència de la PCR convencional, això permet evitar l'ús posterior de tècniques d'electroforesi per visualitzar els resultats de totes les mostres. Malgrat que la PCR quantitativa és una tècnica cinètica, sol ser avaluada a punt final. Així, aquesta tècnica porta a l'obtenció de resultats més ràpids, i/o una menor despesa de reactius emprats en les tècniques d'electroforesi, si segons el criteri de l'investigador, posteriorment cal córrer en gels només les mostres, els resultats previs al PCR en temps real es poden considerar dubtosos i/o per ratificar resultats en mostres positives.
Quantificació de l'expressió gènica[modifica]
La quantificació es pot realitzar en termes absoluts o relatius. En el primer cas, l'estratègia és relacionar el senyal d'amplificació obtingut amb el contingut en ADN emprant una corba de calibratge; és vital que les PCR de la mostra i dels elements de la recta de calibratge tinguin la mateixa eficiència d'amplificació. En el segon cas, s'expressa el canvi en els nivells d'expressió d'ARN missatger (ARNm) interpretat com a ADN complementari (ADNc, generat per retrotranscripció de l'ARNm); aquesta quantificació relativa és més fàcil de realitzar, car no requereix la corba de calibratge, i es basa en la comparació entre el nivell d'expressió del gen estudiat versus un gen de control (també anomenat de referència, intern o normalitzador i, en anglès, housekeeping gene).
Per tant, en la quantificació relativa és irrellevant en quines unitats s'expressa la quantificació i els seus resultats són comparables entre múltiples experiments de RT-Q-PCR. De fet, el propòsit d'utilitzar un o més gens de normalització és corregir la variació no específica, com diferències en la quantitat i qualitat de l'ARN emprat, que poden afectar les eficiències de retrotranscripció i de PCR. No obstant això, l'aspecte crucial és que l'estabilitat del gen de referència sigui una realitat.[15]
La selecció dels gens interns s'ha realitzat clàssicament en biologia molecular analitzant l'estabilitat de l'expressió en estudis qualitatius o de baixa sensibilitat, com l'examen visual de gels d'ARN, densitometria de Northern blots o PCR semiquantitativa (PCR Mimic). En plena era de la genòmica, és possible realitzar una aproximació a gran escala utilitzant els xips d'ADN en el cas de molts organismes.[16] Tanmateix, s'ha observat que la majoria dels gens empleats com normalitzadors en la quantificació de l'expressió d'ARN missatger varien segons les condicions experimentals.[17][18] Per això, cal fer un estudi metodològic previ emprant eines estadístiques per tal de seleccionar els gens més apropiats.[19]
S'han desenvolupat diversos algorismes estadístics que detecten quin o quins gens són els més apropiats per normalitzar un conjunt de teixits sota unes condicions donades. Alguns, com geNORM o BestKeeper, realitzen sobre una matriu d'expressió de gens de referència per diversos teixits, comparacions per parells i mitjanes geomètriques.[7][20]
Modelització[modifica]
A diferència de la PCR de punt final (PCR convencional), la PCR en temps real permet quantificar el nivell de producte obtingut a qualsevol moment de l'amplificació mitjançant el senyal de fluorescència o, més concretament, mitjançant el seu nivell sobre un llindar. Els valors de fluorescència són expressats en forma de logaritmes per tal d'estudiar fàcilment la fase exponencial d'amplificació, que apareix com una línia recta en representar gràficament el logaritme de la fluorescència davant del nombre de cicle. Aquest segment, denominat segment quantificable, permet valorar la quantitat d'ADN inicial.
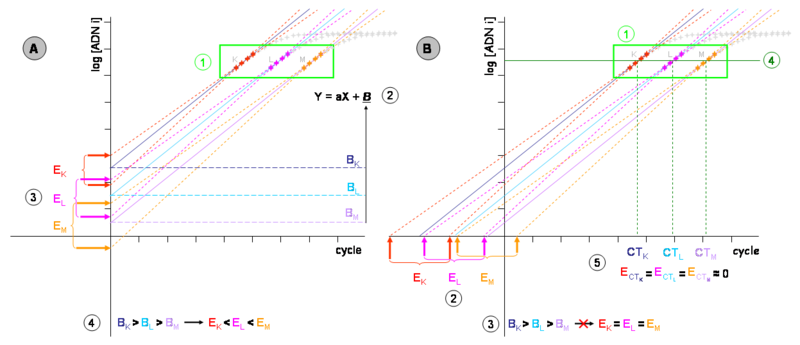
- Gràfic de l'esquerra (A)
Les cinètiques de tres PCR de tres mostres diferents (K, L i M) de concentracions d'ADN inicial decreixents (concretament, cada vegada es redueixen a 1/10) es representen en una gràfica logarítmica de la fluorescència (en ordenades) davant del cicle de PCR (en abscisses). No es representen les fases prèvies a l'exponencial.
- Zona de segments quantificables de cada mostra. La zona de saturació, fora ja de la zona lineal, es mostra en gris.
- Cadascun dels segments quantificables permet definir una equació de la recta de tipus U = ax b, que permet modelitzar l'eficiència de l'amplificació (el pendent a) i, mitjançant l'ordenada a l'origen b, la quantitat d'ADN al cicle 0, almenys teòricament.
- Com que les mesures experimentals comporten un error, aquesta quantificació arrossega un error estocàstic. Si les mostres de concentracions K, L i M fossin amplificades durant més cicles, s'obtindrien cinètiques diferents, tot i que bastant properes (se'n representen els segments quantificables en vermell, rosa i taronja). Cadascun d'aquestes mesuraments permet establir una nova equació, representada per punts del mateix color. Les ordenades a l'origen b diferents també són mesuraments. Així, es pot valorar l'error o la incertesa de la tècnica avaluant les diferències entre els pendents b (EK, EL, EM).
- No obstant això, aquesta imprecisió en el mesurament varia per cada mostra (MK és inferior a EL, que al seu torn és més petita que EM). Una projecció de l'equació de la dreta sobre l'eix d'ordenades o una de les seves paral·leles dona lloc a un "error dependent de la concentració d'ADN inicial".
- Gràfic de la dreta (B)
- Les equacions obtingudes segons els segments quantificables es poden extrapolar a l'eix d'abscisses, tot i que representen un valor sense significat bioquímic, simplement matemàtic.
- Els angles que modelitzen l'error experimental (línies de punts vermells, taronges i roses) permeten definir les noves incerteses EK, EL i EM. Aquestes incerteses són més grans que al gràfic de l'esquerra (gràfic A) tot i mantenir la proporcionalitat. Per tant, es genera un "error independent de la concentració d'ADN inicial" en el mesurament.
- És possible projectar rectes paral·leles sobre la recta definida sobre una paral·lela a l'eix d'abscisses de manera que es tallin els segments quantificables per la meitat. Aquest segment és denominat "llindar de detecció".
- Els valors en X (nombre de cicles) d'aquestes interseccions són anomenats "CT" (o "Ct", de l'anglès cycle threshold, "cicle llindar") o "CP" (de l'anglès crossing point, "punt de creuament"). Es tracta dels valors matemàtics definits sobre l'espai dels reals positius i no dels enters positius (encara que una fracció del cicle no tingui realitat experimental). Aquests valors inversament proporcionals a la quantitat d'ADN inicial solen presentar una incertesa mínima sobre el mesurament, generalment de menys d'un 5%.
Rectes de calibratge[modifica]
L'ús del CT com a valor matemàtic permet obtenir resultats fiables, però això no es pot aprofitar directament. Per tal de conèixer la quantitat d'ADN inicial, cal realitzar noves transformacions matemàtiques que requereixen conèixer l'eficiència de la PCR, que se sol determinar gràcies a una recta de calibratge.

- Gràfic de l'esquerra (A)
Les noves mostres F, G, H, I, J, K, L, M i N de concentració d'ADN inicial decreixent (un ordre de magnitud cada vegada) són amplificades mitjançant la PCR en un mateix experiment. Cada cinètica permet determinar un CT per cadascuna (és a dir, un número en referència a un cicle en concret). Es representen les concentracions d'ADN en nombre de molècules per tub (en aquest cas, F en té 70 milions i N en té 0,7).
Els rendiments representats corresponen a n reaccions. La fiabilitat és deguda a l'existència de rèpliques independents a la reacció, el personal i els reactius. La fluorescència es mostra en unitats arbitràries i el soroll de fons ha estat netejat. Cal destacar que la mostra Nn no s'ha amplificat gens.
- Gràfic de la dreta (B)
Els valors CT mitjans en funció de la quantitat d'ADN inicial de totes les rèpliques es poden representar en un gràfic semilogarítmic. D'aquesta manera, la recta de calibratge per aquests valors mitjans es pot definir gràcies a una regressió lineal amb un determinat coeficient de correlació (r2) que, per ser considerat de qualitat, ha de tenir un valor de 0,9999. No obstant això, s'han de representar els errors, generalment definits segons el rang de valors obtinguts per un punt.
Amb aquestes dades, és possible estudiar:
- La fase quantitativa i detectable de la PCR, en què totes les mostres són detectables i s'alineen en tots els seus punts amb la seva recta particular. Generalment, aquesta fase comprèn les concentracions d'ADN inicials al voltant de 102 o 108 còpies de l'àcid nucleic. Per sota d'aquestes xifres, els fenòmens estocàstics alteren perceptiblement els resultats, tot i que és possible compensar-ho amb un nombre elevat de rèpliques als mesuraments. Per sobre d'aquests valors, el "soroll de fons" és prou baix per no poder ser determinat; en aquest cas, és possible compensar aquesta deficiència mitjançant un protocol de PCR molt eficient o, de manera més senzilla, diluint la mostra inicial.
- La fase a vegades detectable però no quantitativa de la PCR, que comprèn les concentracions d'ADN inicial entre una còpia i desenes de còpies. En l'exemple del gràfic, el percentatge de mostres mesurades s'indica mitjançant les concentracions M i N, és a dir, 83% per una concentració mitjana de set còpies i 28% quan tres dels quatre tubs contenen una còpia (0,7 de concentració, o -0,15 en logaritme). La dispersió de les mesures, i per tant el marge d'error, augmenten perceptiblement. S'observa que un bon nombre de mesures M i N no s'alineen amb la seva recta particular, encara que no ho facin per motius aleatoris. Per tant, només cal replicar més vegades els mesuraments per determinar amb precisió aquesta fase.
- La recta de calibratge representada en un gràfic semilogarítmic dona lloc a una recta definida per la fórmula I = aX + B, on:
- Y és el CT mesurat pel termociclador.
- El pendent a deriva de l'eficiència de la PCR, i pot ser calculada mitjançant l'equació
En l'esquema es representa el cas més freqüent, en què la concentració està expressada en logaritmes decimals. Aquest pendent és sovint considerat com una constant d'amplificació per cada gen, parell d'encebadors i condició de PCR particular. Per això s'utilitza aquest pendent o eficiència d'amplificació per quantificar en la PCR quantitativa.
- X és la concentració d'ADN inicial expressada en logaritme de còpies/tub, ng/ml, unitats arbitràries, etc.
- B és un punt matemàtic sense realitat experimental (log 0 = 1/∞) que pot ser emprat per comparar cada experiment de PCR amb experiments similars. D'aquesta manera, si les rectes de calibratge ho han estat per la seva intersecció a l'origen B, la dispersió serà menor, excepte pels punts M i N.
- La recta de regressió no passa pel punt central de la dispersió en cada mesurament per les concentracions M i N, però sí segons el pendent i un factor d'amortiment. És a dir, si es consideren CT mitjans per L i N, seran modelitzats de millor forma mitjançant un polinomi de segon grau. El programari d'alguns termocicladors permet tenir en compte aquest amortiment, tot i que cal tenir en compte que:
- Aquest amortiment pot variar dràsticament d'una recta de calibratge a l'altra, de manera que la correcció que es modelitza probablement no correspon a la que passa per la mostra quantificada. El programari comercial ho modelitza per una sola mostra.
- La imprecisió en la quantificació en aquestes concentracions és tan important que pot resultar necessari modelitzar l'amortiment.
- Quan es dibuixen les rectes, aquest amortiment correspon a una reducció del pendent després d'un augment de l'eficàcia de la PCR, reduint els efectes estocàstics. També s'observa que aquest amortiment es defineix principalment segons la concentració més baixa (N, 0,7 còpies a l'exemple), és a dir, no podrà realitzar cap amplificació si no es té almenys una molècula completa de l'ADN per amplificar. El biaix produït en la distribució gaussiana de l'error pot provocar aquesta aparició de dades estranyes o amortiments mitjans.
La recta de calibratge permet una quantificació per un protocol experimental donat, però cal tenir en compte que existeixen moltes fonts d'errors potencials, com ara diferències de composició química de l'amortidor de reacció, de les mostres (presència de proteïnes, ARN, etc.) i fins i tot del diluent (generalment aigua).
Enfocaments[modifica]
És possible quantificar l'expressió gènica tant en termes absoluts com en terme relatius (és a dir, en proporció a l'expressió d'un altre gen). En el segon cas és molt important seleccionar com a gen estàndard un gen del qual que l'expressió no variï quan la mostra objecte de l'anàlisi se sotmet experimentalment a diferents condicions (per exemple, un estrès ambiental[21] o biòtic[22] o cel·lular[23]).
- Quantificació absoluta
Es basa en les característiques de la fase exponencial de la corba sigmoide d'emissió de fluorescència.[24] Segueix l'equació:
- on Q és la quantitat d'ADN, n. correspon al nombre de cicle, o és el cicle de partida i E l'eficiència de la reacció. Al cicle en què se sobrepassa el nivell llindar (és a dir, el CT), es diu que:
(1)
- Quantificació relativa
S'estableix una relació R entre la quantitat d'ADN inicial d'una mostra respecte a la d'un testimoni, que s'expressa de forma invariable i independent del tractament. En la seva detecció s'observa que:[8]
- Substituint de (1) s'obté que
- i, per tant,
o:
Des del 2002 es tendeix a emprar diversos gens normalitzadors per fer una quantificació relativa fiable; d'aquesta manera, es generen factors de normalització que ponderen d'alguna manera l'impacte de cada gen intern. Una aproximació comuna es basa en l'ús de mitjanes geomètriques d'aquests gens normalitzadors.[7]
Selecció de gens de normalització[modifica]
L'anàlisi estadística que permet detectar els gens expressats de forma més estable en la sèrie de teixits estudiada és crucial, car en depèn l'elecció dels gens utilitzats com a control intern durant els assajos de quantificació relativa. Per avaluar aquesta estabilitat, en alguns casos se'n comparen els valors CT, i en altres les quantitats relatives emprant la mostra amb el CT més baix com a element normalitzador.
Com a primera aproximació, l'algorisme geNorm permet calcular un valor d'estabilitat M inversament proporcional a l'estabilitat del gen en qüestió, així com una sèrie de valors PV que defineixen el nombre mínim de gens adequats per efectuar una normalització fiable.[7] El còmput del valor M es basa en la variació mitjana, calculada comparant parells de dos en dos, d'un gen respecte als altres gens presents a l'estudi. L'algorisme es calcula en diversos passos, car a cada ronda de comparacions se suprimeix el gen que ha obtingut un pitjor valor. D'aquesta manera, es pot adjudicar un valor M a cada gen tret dels més estables, que comparteixen el mateix valor. El còmput dels valors PV es basa en el disseny d'un factor de normalització òptim, que al principi es realitza amb els dos gens més estables, però posteriorment se li'n afegeixen altres fins que aquest afegit no en millora significativament la precisió. Així doncs, es defineix un valor PV per les comparacions entre Vn/n1, estimat com la desviació estàndard dels ràtios transformats logarítmicament de . L'any 2009 fou publicada una actualització de geNorm especialment útil per normalitzar les dades provinents d'un conjunt ampli i sense biaix de gens, com -per exemple- els perfils d'expressió dels microARNs.[25]
Un altre algorisme anomenat NormFinder aprofita els valors CT per ajustar un model matemàtic de l'expressió gènica que sigui capaç d'avaluar les variacions presents dins i entre els grups (tipus d'òrgans, tractaments químics, etc.) definits per l'investigador. D'aquesta manera, els gens amb una variació inter i intragrup mínima són considerats com els més estables, amb un valor d'estabilitat mínim S.[26]
També és possible analitzar el coeficient de variació dels nivells d'expressió relatius normalitzats. Aquesta aproximació, pròpia de qBase o qBasePlus,[27] requereix transformar els valors CT inicials en quantitats relatives tenint en compte els valors d'eficiència de la PCR i emprant com a calibrador el gen amb el CT més baix. Seguidament, es calcula un factor de normalització per cada mostra emprant la mitjana geomètrica dels valors relatius estimats per tots els gens candidats.
Ús de sondes específiques[modifica]
La PCR en temps real es pot fer marcant amb fluorescència oligonucleòtids que detecten específicament l'aparició del producte desitjat. Aquesta tècnica es basa en l'ús del FRET o transmissió d'energia de ressonància, que és un mecanisme de transferència d'energia entre cromòfors. El FRET es fonamenta en el fet que l'excitació d'un cromòfor es pot transferir a un altre de proper, generalment quan tots dos es troben a la mateixa molècula, mitjançant un mecanisme acoblador dipol-dipol.[28] En el cas que els cromòfors siguin fluorescents (fluorocroms), el mecanisme subjacent continua sent el mateix: l'energia es transfereix, cosa que provoca l'aparició de fluorescència (la fluorescència en si no és transferida)[29][30] Les sondes emprades en PCR en temps real són:

- Les sondes TaqMan permeten mesurar la producció de productes de PCR mitjançant un sistema de sondes marcades amb dos fluorocroms. La seva utilitat és que posseeixen un fluoròfor a l'extrem 3' i una molècula al 5' que bloqueja la seva emissió de fluorescència (denominada en anglès quench); aquesta sonda marcada hibrida específicament a la part central del producte de PCR per obtenir. D'aquesta manera, en fer la PCR (amb la sonda i el parell d'encebadors específics), la sonda hibrida a l'amplicó, però, a causa de la proximitat del fluoròfor a quench, no s'emet fluorescència; quan la polimerasa topa amb la sonda la hidrolitza mitjançant la seva activitat exonucleasa 5'- 3', provocant la separació del quench del fluorocrom i, per tant, l'emissió de fluorescència proporcional a la quantitat d'amplicó produït.[31]

- Les sondes del tipus balisa molecular[32] també són oligonucleòtids de cadena simple que, per la seva estructura, tenen una zona d'aparellament de bases interna i, per tant, formen una forqueta. En presència de l'amplicó la sonda s'obre i s'hi uneix preferentment, cosa que produeix l'emissió de fluorescència. Estructuralment, tenen la zona complementària a l'amplicó al gir de la forqueta, la de complementarietat interna al coll i els fluoròfors als extrems: el donador en el 5' i l'acceptor en el 3'. Quan la sonda està tancada en forqueta, el fluoròfor del 3' impedeix l'emissió de fluorescència per part del de l'extrem 5', cosa que no passa en unir-se a l'amplicó. Aquestes sondes són de gran utilitat per detectar polimorfismes d'un sol nucleòtid en estudis farmacogenòmics.[33] S'empren també per filiar amb exactitud agents infecciosos de difícil tipificació amb altres mètodes i pel control quantitatiu de la càrrega vírica en determinats malalts.[34]

- Les sondes Scorpion són molècules mixtes contenint un encebador específic per l'amplicó, unit covalentment a una forqueta similar a les sondes del tipus balisa molecular que tenen un element complementari a l'amplicó a la zona de gir.[35] Els fluoròfors donador i acceptor es troben a l'estructura de forqueta, de manera que quan està tancada no es produeix l'emissió de fluorescència, cosa que sí que passa quan tots dos se separen a causa de la presència de l'amplicó i l'obertura de la zona en forqueta.[36]
Aplicacions tecnològiques[modifica]
Tot i que l'aplicació més estesa de la Q-PCR és l'avaluació de l'expressió gènica de gens concrets de forma relativa (emprant ARNm de la mostra i retrotranscrivint-lo a ADNc, que es mesura mitjançant la tècnica), se n'han desenvolupat altres fora de l'entorn purament acadèmic. Les aplicacions rellevants per la indústria inclouen la quantificació de càrrega microbiana en aliments o en material vegetal, la detecció d'OGM (organismes genèticament modificats) i la quantificació i genotipatge de patògens vírics en humans.
Detecció de fitopatògens[modifica]
La producció de propàguls vegetals o plàntules lliures de patògens és una constant en la indústria agronòmica per qüestions econòmiques i de sanitat. En el cas de Phytophthora ramorum, un fong causant de mort sobtada en roures i altres espècies, s'han desenvolupat sistemes basats en sondes TaqMan que són capaços de detectar una quantitat de 10 a 100 fg barrejats a l'ADN de la planta hoste. La discriminació entre l'ADN del patogen i de la planta es fa amplificant seqüències ITS, uns espaiadors situats a la zona codificant dels gens d'ARN ribosòmics que són característics de cada tàxon.[37] N'existeixen variants amb sondes scorpion, balises moleculars i LAMP pel mateix patogen, que també, poden ser aplicades al camp.[38]
Detecció d'OGM[modifica]
La Q-PCR (precedida per una retrotranscripció) s'empra en la detecció d'OGM per la seva alta sensibilitat i rang dinàmic de detecció d'ADN. Les alternatives com l'anàlisi de l'ADN o de les proteïnes solen tenir menys sensibilitat. Per això s'empren encebadors específics que amplifiquen no el mateix transgèn, sinó el seu promotor, terminador i fins i tot les seves seqüències intermèdies, emprades durant el procés d'enginyeria del vector. A més, com que el procés de creació d'una planta transgènica sol conduir a la inserció de més d'una còpia del transgèn, cal avaluar la seva quantitat. Per fer-ho, es duu a terme una quantificació relativa emprant com a gen control un que sigui propi de l'espècie tractada i que es trobi en còpia única.[39][40]
Quantificació i genotipatge en medicina clínica[modifica]
Les virosis humanes poden ser degudes a infeccions per part d'un únic agent patogen o ser el resultat d'una coinfecció per diferents virus, fet que dificulta el seu diagnòstic mitjançant les tècniques clàssiques, complicant alhora l'elecció del tractament adequat i el pronòstic de la malaltia. L'ús de la Q-PCR possibilita tant la quantificació com el genotipatge (caracterització de la soca mitjançant corbes de fusió) de virus com l'HBV (virus de l'hepatitis B)[41] o el virus d'Ebola.[42] El grau d'infecció, quantificat en còpies de genoma víric per unitat de teixit del pacient, és important en molts casos; per exemple, la probabilitat que es reactivi el virus de l'herpes simple de tipus 1, està relacionada amb el nombre de neurones infectades als ganglis.[43] Aquesta quantificació es realitza amb retrotranscripció o sense, en el cas que els virus s'integrin al genoma humà en algun moment del seu cicle, com ocorre en les infeccions per determinats virus del papil·loma humà (VPH-16 i 18) associades amb l'aparició de càncer de cèrvix[44] i d'orofaringe.[45]
Referències[modifica]
- ↑ (de l'anglès quantitative polymerase chain reaction)
- ↑ (de l'anglès real time PCR)
- ↑ Watson, J. D.; Baker, T. A.; Bell, S. P.; Gann, A.; Levine, M. i Losick, R. Molecular Biology of the Gene. 5a ed.. San Francisco: Benjamin Cummings, 2004. ISBN 0-321-22368-3.
- ↑ (RT-PCR, de l'anglès reverse transcriptase PCR),
- ↑ Freeman WM, Walker SJ, Vrana KE «Quantitative RT-PCR: pitfalls and potential» (en anglès). Biotechniques, 1999 Gen; 26 (1), pp: 112-22, 124-5. DOI: 10.2144/99261rv01. ISSN: 1940-9818. PMID: 9894600 [Consulta: 7 octubre 2019].
- ↑ 6,0 6,1 Michael W. Pfaff, Ales Tichopad, Christian Prgomet i Tanja P. Neuvians (2005). Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper – Excel-based tool using pair-wise correlations Arxivat 2019-09-13 a Wayback Machine. Biotechonology Letters 26:509-515
- ↑ 7,0 7,1 7,2 7,3 Vandesompele J, De Preter K, Pattyn F, Poppe B, Van Roy N, De Paepe A, Speleman F «Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes» (en anglès). Genome Biol, 2002 Jun 18; 3 (7), pp: RESEARCH0034 [Consulta: 8 octubre 2019].
- ↑ 8,0 8,1 Pfaffl MW, Horgan GW, Dempfle L (2002). Relative expression software tool (REST©) for group-wise comparison and statistical analysis of relative expression results in real-time PCR. Nucl. Acids Res. 30: e36. ISSN 1362-4962 doi:10.1093/nar/30.9.e36 PMID: 11972351
- ↑ Rychlik W, Spencer WJ, Rhoads RE «Optimization of the annealing temperature for DNA amplification in vitro». Nucl Acids Res, 18 (21), 1990 Nov 11, pàg. 6409-6412. DOI: 10.1093/nar/18.21.6409. PMC: 332522. PMID: 2243783.
- ↑ Joseph Sambrook i David W. Russel. Molecular Cloning: A Laboratory Manual. 3a edició. Cold Spring Harbor (Nova York): Cold Spring Harbor Laboratory Press, 2001. ISBN 0-87969-576-5.
- ↑ PubChem «SYBR Green I» (en anglès). Compound Summary. National Center for Biotechnology Information, US National Library of Medicine, 2019 Oct 5; CID 10436340 (rev), pàgs: 9 [Consulta: 6 octubre 2019].
- ↑ Zipper H, Brunner H, Bernhagen J, Vitzthum F «Investigations on DNA intercalation and surface binding by SYBR Green I, its structure determination and methodological implications» (en anglès). Nucleic Acids Res, 2004 Jul 12; 32 (12), pp: e103. DOI: 10.1093/nar/gnh101. PMC: 484200. PMID: 15249599 [Consulta: 6 octubre 2019].
- ↑ Ponchel F, Toomes C, Bransfield K, Leong FT, et al «Real-time PCR based on SYBR-Green I fluorescence: an alternative to the TaqMan assay for a relative quantification of gene rearrangements, gene amplifications and micro gene deletions» (en anglès). BMC Biotechnol, 2003 Oct 13; 3 (1), pp: 18. DOI: 10.1186/1472-6750-3-18. PMC: 270040. PMID: 14552656 [Consulta: 6 octubre 2019].
- ↑ Ririe KM, Rasmussen RP, Wittwer CT «Product Differentiation by Analysis of DNA Melting Curves during the Polymerase Chain Reaction» (en anglès). Anal Biochem, 1997 Feb 15; 245 (2), pp: 154-160. DOI: 10.1006/abio.1996.9916. ISSN: 1096-0309. PMID: 9056205 [Consulta: 8 octubre 2019].
- ↑ Brunner AM, Iàkovlev IA, Strauss SH «Validating internal controls for quantitative plant gene expression studies» (en anglès). BMC Plant Biol, 2004 Ag 18; 4, pp: 14. DOI: 10.1186/1471-2229-4-14. PMC: 515301. PMID: 15317655 [Consulta: 7 octubre 2019].
- ↑ Czechowski T, Stitt M, Altmann T, Udvardi MK, Scheible WR «Genome-wide identification and testing of superior reference genes for transcript normalization in Arabidopsis» (en anglès). Plant Physiol, 2005 Set; 139 (1), pp: 5-17. DOI: 10.1104/pp.105.063743. PMC: 1203353. PMID: 16166256 [Consulta: 8 octubre 2019].
- ↑ Thellin O, Zorzi W, Lakaye B, De Borman B, et al «Housekeeping genes as internal standards: use and límits» (en anglès). J Biotechnol, 1999 Oct 8; 75 (2-3), pp: 291-295. DOI: 10.1016/s0168-1656(99)00163-7. ISSN: 1873-4863. PMID: 10617337 [Consulta: 6 octubre 2019].
- ↑ Dheda K, Huggett JF, Bustin SA, Johnson MA, Rook G, Zumla A (2004). Validation of housekeeping genes for normalizing RNA expression in real-time PCR. Biotechniques 37 (1), pp: 112-119. PMID: 15283208 doi:10.2144/04371RR03 ISSN 1940-9818
- ↑ Radonic A, Thulke S, Mackay IM, Landt O, Siegert W, Nitsche A «Guideline for reference gene selection for quantitative real-time PCR» (en anglès). Biochem Biophys Res Commun, 2004 Gen 23; 313 (4), pp: 856-862. DOI: 10.1016/j.bbrc.2003.11.177. ISSN: 1090-2104. PMID: 14706621 [Consulta: 8 octubre 2019].
- ↑ Pfaffl MW, Tichopad A, Prgomet C, Neuvians TP (2004). Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper—Excel-based tool using pair-wise correlations. Biotechnol Lett 26 (6), pp: 509-515. PMID: 15127793 ISSN 1573-6776
- ↑
- ↑ Nicot, Nathalie; Hausman, Jean-Francois; Hoffmann, Lucien; Evers, Daniele «Housekeeping gene selection for real-time RT-PCR normalization in potato during biotic and abiotic stress». Journal of Experimental Botany, 56, 421, 2005, p. 2907-2914. DOI: 10.1093/jxb/eri285.
- ↑ McLoughlin KJ, Pedrini E, MacMahon M, Guduric-Fuchs J, Medina RJ «Selection of a Real-Time PCR Housekeeping Gene Panel in Human Endothelial Colony Forming Cells for Cellular Senescence Studies» (en anglès). Front Med (Lausanne), 2019 Mar 11; 6, pp: 33. DOI: 10.3389/fmed.2019.00033. PMC: 6421261. PMID: 30915334 [Consulta: 3 novembre 2019].
- ↑ Bustin, S.A. «Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays». Journal of Molecular Endocrinology, 25, 2, 2000, p. 169-193. DOI: 10.1677/jme.0.0250169.
- ↑ Mestdagh P, Van Vlierberghe P, De Weer A, Muth D, et al «A novel and universal method for microRNA RT-qPCR data normalization» (en anglès). Genome Biol, 2009; 10 (6), pp: R64. DOI: 10.1186/gb-2009-10-6-r64. PMC: 2718498. PMID: 19531210 [Consulta: 3 novembre 2019].
- ↑ Andersen, Claus Lindbjerg; Jensen, Jens Ledet; Orntoft, Torben Falck «Normalization of Real-Time Quantitative Reverse Transcription-PCR Data: A Model-Based Variance Estimation Approach to Identify Genes Suited for Normalization, Applied to Bladder and Colon Cancer Data Sets» (w). Cancer Research, 64, 15, 2004, p. 5245-5250. DOI: 10.1158/0008-5472.CAN-04-0496.
- ↑ Hellemans, J.; Mortier, G.; De Paepe, A.; Speleman, F. «qBase relative quantification framework and software for management and automated analysis of real-time quantitative PCR data». Genome Biol, 8, 2, 2007, p. R19. DOI: 10.1186/gb-2007-8-2-r19.
- ↑ Förster, T «Zwischenmolekulare Energiewanderung und Fluoreszenz» (en alemany). Ann Phys, 1948; 437 (1-2), pp: 55-75. DOI: 10.1002/andp.19484370105. ISSN: 1521-3889 [Consulta: 14 octubre 2019].
- ↑ Lakowicz, JR «Principles of Fluorescence Spectroscopy (Third Edition)» (en anglès). Springer Science+Business Media, LLC, 2006, pàgs: 960. ISBN 978-0387-31278-1 [Consulta: 14 octubre 2019].
- ↑ Tutorial de microscòpia FRET a Olympus (anglès)
- ↑ Heid, C.A.; Stevens, J.; Livak, K.J.; Williams, P.M. «Real time quantitative PCR». Genome Research, 6, 10, 1996, p. 986-994. DOI: 10.1101/gr.6.10.986.
- ↑ Tyagi S, Kramer FR «Molecular beacons: probes that fluoresce upon hybridization» (en anglès). Nat Biotechnol, 1996 Mar; 14 (3), pp: 303-308. DOI: 10.1038/nbt0396-303. ISSN: 1546-1696. PMID: 9630890 [Consulta: 7 octubre 2019].
- ↑ Mhlanga MM, Malmberg L «Using molecular beacons to detect single-nucleotide polymorphisms with real-time PCR» (en anglès). Methods, 2001 Des; 25 (4), pp: 463-471. DOI: 10.1006/meth.2001.1269. ISSN: 1046-2023. PMID: 11846616 [Consulta: 7 octubre 2019].
- ↑ Abravaya K, Huff J, Marshall R, Merchant B, et al «Molecular beacons as diagnostic tools: technology and Applications» (en anglès). Clin Chem Lab Med, 2003 Abr; 41 (4), pp: 468-474. DOI: 10.1515/CCLM.2003.070. ISSN: 1437-4331. PMID: 12747588 [Consulta: 2 novembre 2019].
- ↑ Carters R, Ferguson J, Gaut R, Ravetto P, et al «Design and use of scorpions fluorescent signaling molecules» (en anglès). Methods Mol Biol, 2008; 429, pp: 99-115. DOI: 10.1007/978-1-60327-040-3_8. ISSN: 1940-6029. PMID: 18695962 [Consulta: 2 novembre 2019].
- ↑ Whitcombe, D.; Theaker, J.; Guy, S.P.; Brown, T. «Detection of PCR products using self-probing amplicons and fluorescence». Nature Biotechnology, 17 (8), 1999; Ag, p. 804-807. DOI: 10.1038/11751. ISSN: 1546-1696.
- ↑ Baldwin, B.G. «Phylogenetic utility of the internal transcribed spacers of nuclear ribosomal DNA in plants: an example from the compositae». Molecular Phylogenetics and Evolution, 1 (1), 1992, pàg. 3-16. DOI: 10.1016/1055-7903(92)90030-K. ISSN: 1055-7903. PMID: 1342921.
- ↑ Tomlinson, J. A.; Barker, I.; Boonham, N. «Faster, Simpler, More-Specific Methods for Improved Molecular Detection of Phytophthora ramorum in the Field» (w). Applied and Environmental Microbiology, 73, 12, 2007, p. 4040-4047. DOI: 10.1128/AEM.00161-07.
- ↑ Holst-jensen, A.; R{o}nning, S.B.; L{o}vseth, A.; Berdal, K.G. «PCR technology for screening and quantification of genetically modified organisms (GMOs)» (w). Analytical and Bioanalytical Chemistry, 375, 8, 2003, p. 985-993.
- ↑ Brodmann PD, Ilg EC, Berthoud H, Herrmann A «Real-time quantitative polymerase chain reaction methods for four genetically modified maize varieties and maize DNA content in food» (en anglès). J AOAC Int, 2002 Maig-Jun; 85 (3), pp: 646-653. DOI: 10.5555/jaoi.2002.85.3.646. ISSN: 1060-3271. PMID: 12083257 [Consulta: 8 octubre 2019].
- ↑ Yeh, S.H.; Tsai, C.Y.; Kao, J.H.; Liu, C.J. «Quantification and genotyping of hepatitis B virus in a single reaction by real-time PCR and melting curve analysis». Journal of Hepatology, 41, 4, 2004, p. 659-666. PMID: 15464248. DOI: 10.1016/j.jhep.2004.06.031. ISSN: 1600-0641.
- ↑ Cherpillod P, Schibler M, Vieille G1, Cordey S, et al «Ebola virus disease diagnosis by real-time RT-PCR: A comparative study of 11 different procedures» (en anglès). J Clin Virol, 2016 Abr; 77, pp: 9-14. DOI: 10.1016/j.jcv.2016.01.017. ISSN: 1873-5967. PMID: 26874083 [Consulta: 4 novembre 2019].
- ↑ Sawtell, N.M. «The probability of in vivo reactivation of herpes simplex virus type 1 increases with the number of latently infected neurons in the ganglia». Journal of Virology, 72, 8, 1998, p. 6888-6892. ISSN: 1098-5514.
- ↑ Peter, M.; Rosty, C.; Couturier, J.; Radvanyi, F. «MYC activation associated with the integration of HPV DNA at the MYC locus in genital tumors». Oncogene, 25, 44, 2006; Maig 8, p. 5985-5993. DOI: 10.1038/sj.onc.1209625. ISSN: 1476-5594.
- ↑ Gao G, Chernock RD, Gay HA, Thorstad WL, et al «A novel RT-PCR method for quantification of human papillomavirus transcripts in archived tissues and its application in oropharyngeal cancer prognosis» (en anglès). Int J Cancer, 2013 Feb 15; 132 (4), pp: 882-890. DOI: 10.1002/ijc.27739. PMC: 3498587. PMID: 22821242 [Consulta: 3 novembre 2019].
Bibliografia[modifica]
- Elyse Poitras i Alain Houde (2002). "La PCR en temps réel: principes et applications". Reviews in Biology and Biotechnology. 2(2):2-11. (francès)
- Bustin S. A. (2000)."Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays". J Mol Endocrinol. 25(2):169-93. (anglès)
- Higuchi, R., Dollinger, G., Walsh, P. S. i Griffith, R. (1992). "Simultaneous amplification and detection of specific DNA-sequences". Bio-Technology 10 (4), 413-417. (anglès)
- Holland, P. M., Abramson, R. D., Watson, R. i Gelfand, D. H. (1991). "Detection of specific polymerase chain reaction product by utilizing the 50 !30 exonuclease activity of Thermus aquaticus DNA polymerase". Proc. Natl. Acad. Sci. USA 88 (16), 7276-7280. (anglès)
- Kubista M., Andrade J. M., Bengtsson M., Forootan A., Jonák J., Lind K., Sindelka R., Sjöback R., Sjögreen B., Strömbom L., Ståhlberg A. i Zoric N. (2006). "The real-time polymerase chain reaction". Mol Aspects Med. 27(2-3):95-125. (anglès)
- Thermo Fisher Scientific. Real-time PCR handbook (en anglès). Lifetechnologies.com, 2014; CO010759 0914, pàgs: 73 [Consulta: 30 octubre 2019].
- Bustin, Stephen; Nolan, Tania. Talking the talk, but not walking the walk: RT-qPCR as a paradigm for the lack of reproducibility in molecular research (en anglès). Eur J Clin Invest, 2017 Oct; 47 (10), pp: 756-774. PMID: 28796277. DOI 10.1111/eci.12801 [Consulta: 3 novembre 2019].
- Ståhlberg, Anders; Kubista, Mikael. Technical aspects and recommendations for single-cell qPCR (en anglès). Mol Aspects Med, 2018 Feb; 59, pp: 28-35. PMID: 28756182. DOI 10.1016/j.mam.2017.07.004 [Consulta: 7 octubre 2019].
Enllaços externs[modifica]
| A Wikimedia Commons hi ha contingut multimèdia relatiu a: PCR en temps real |
- Q-PCR Academic & Industrial Information Platform (anglès)
- Tutorial sobre la Q-PCR al web de la Universitat de Carolina del Sud (anglès)
- The principle of Real Time PCR, Reverse Transcription, quantitative rt-PCR Biomedical and Biological Sciences. 2016 Des (anglès)
- PCR en tiempo real (qPCR): Conceptos Básicos Ortiz Casas, B. 2018 Set (castellà)
- PCR en temps réel FAO. 2012 Jun (francès)

![{\displaystyle E=(index\;del\;logaritme)^{\frac {-1}{pendent}}={\sqrt[{-pendent}]{index\;del\;logaritme}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/d04f105c80251d6be3424fdb0fc0c6141a26e91d)






