ARN de transferència: diferència entre les revisions
#viquiestirada |
Robot posa data a plantilles de manteniment |
||
| Línia 11: | Línia 11: | ||
== Anticodó == |
== Anticodó == |
||
Un '''anticodó'''<ref>{{cite journal|vauthors=Felsenfeld G, Cantoni GL|title=Use of thermal denaturation studies to investigate the base sequence of yeast serine sRNA|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=51|issue=5|pages=818–26|date=May 1964|pmid=14172997|pmc=300168|doi=10.1073/pnas.51.5.818|bibcode=1964PNAS...51..818F}}</ref> és una unitat formada per tres [[Nucleòtid|nucleòtids]] que corresponen a les tres bases del [[codó]] en el [[mRNA]]. Cada [[tRNA]] conté una seqüència de triplet anticodó diferent que pot formar 3 [[parells de bases complementàries]] a un o més codons per a un aminoàcid. Alguns anticodons poden combinar-se amb més d’un codó a causa d’un fenomen conegut com a [[aparellament de bases wobble]]. Sovint, el primer nucleòtid de l’anticodó no es troba a l’ARNm: la [[inosina]], que pot unir [[hidrogen]] a més d’una base en la posició del codó corresponent.<ref name="Stryer2002" /> En el [[codi genètic]], és freqüent que un sol aminoàcid sigui especificat per les quatre possibilitats de tercera posició, o almenys per les [[Pirimidina|pirimidines]] i les [[Purina|purines]]; per exemple, l'aminoàcid [[glicina]] està codificat per les seqüències de codons GGU, GGC, GGA i GGG. Altres nucleòtids modificats també poden aparèixer a la primera posició anticodon, de vegades coneguda com la "posició de balanceig", que produeixen canvis subtils al codi genètic, com per exemple en els [[Mitocondri|mitocondris]].<ref>{{cite journal|vauthors=Suzuki T, Suzuki T|title=A complete landscape of post-transcriptional modifications in mammalian mitochondrial tRNAs|journal=Nucleic Acids Research|volume=42|issue=11|pages=7346–57|date=June 2014|pmid=24831542|pmc=4066797|doi=10.1093/nar/gku390}}</ref> Per cèl·lula, es requeririen 61 tipus de tRNA per proporcionar una correspondència individual entre molècules de tRNA i codons que especifiquen aminoàcids, ja que hi ha 61 codons de sentit del codi genètic estàndard. Tanmateix, moltes cèl·lules contenen menys de 61 tipus de tRNA perquè la base ondulada és capaç de lligar-se a diversos codons que, encara que no necessàriament a tots, especifiquen un determinat aminoàcid. Cal traduir, sense ambigüitat, els 61 codons de sentit, amb un mínim de 31 tRNA; el màxim observat és de 41.<ref name="crick" /><ref>Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J. (2004). ''Molecular Biology of the Cell''. WH Freeman: New York. 5th ed.{{ISBN?}}{{ |
Un '''anticodó'''<ref>{{cite journal|vauthors=Felsenfeld G, Cantoni GL|title=Use of thermal denaturation studies to investigate the base sequence of yeast serine sRNA|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=51|issue=5|pages=818–26|date=May 1964|pmid=14172997|pmc=300168|doi=10.1073/pnas.51.5.818|bibcode=1964PNAS...51..818F}}</ref> és una unitat formada per tres [[Nucleòtid|nucleòtids]] que corresponen a les tres bases del [[codó]] en el [[mRNA]]. Cada [[tRNA]] conté una seqüència de triplet anticodó diferent que pot formar 3 [[parells de bases complementàries]] a un o més codons per a un aminoàcid. Alguns anticodons poden combinar-se amb més d’un codó a causa d’un fenomen conegut com a [[aparellament de bases wobble]]. Sovint, el primer nucleòtid de l’anticodó no es troba a l’ARNm: la [[inosina]], que pot unir [[hidrogen]] a més d’una base en la posició del codó corresponent.<ref name="Stryer2002" /> En el [[codi genètic]], és freqüent que un sol aminoàcid sigui especificat per les quatre possibilitats de tercera posició, o almenys per les [[Pirimidina|pirimidines]] i les [[Purina|purines]]; per exemple, l'aminoàcid [[glicina]] està codificat per les seqüències de codons GGU, GGC, GGA i GGG. Altres nucleòtids modificats també poden aparèixer a la primera posició anticodon, de vegades coneguda com la "posició de balanceig", que produeixen canvis subtils al codi genètic, com per exemple en els [[Mitocondri|mitocondris]].<ref>{{cite journal|vauthors=Suzuki T, Suzuki T|title=A complete landscape of post-transcriptional modifications in mammalian mitochondrial tRNAs|journal=Nucleic Acids Research|volume=42|issue=11|pages=7346–57|date=June 2014|pmid=24831542|pmc=4066797|doi=10.1093/nar/gku390}}</ref> Per cèl·lula, es requeririen 61 tipus de tRNA per proporcionar una correspondència individual entre molècules de tRNA i codons que especifiquen aminoàcids, ja que hi ha 61 codons de sentit del codi genètic estàndard. Tanmateix, moltes cèl·lules contenen menys de 61 tipus de tRNA perquè la base ondulada és capaç de lligar-se a diversos codons que, encara que no necessàriament a tots, especifiquen un determinat aminoàcid. Cal traduir, sense ambigüitat, els 61 codons de sentit, amb un mínim de 31 tRNA; el màxim observat és de 41.<ref name="crick" /><ref>Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J. (2004). ''Molecular Biology of the Cell''. WH Freeman: New York. 5th ed.{{ISBN?}}{{pàgina necessària|data=setembre de 2019}}</ref> |
||
== Gens del tRNA == |
== Gens del tRNA == |
||
Revisió del 03:34, 9 set 2019

L'ARN de transferència (ARNt o tRNA) és un tipus d'ARN que transporta un aminoàcid concret cap als ribosomes a la síntesi de proteïnes durant la traducció. Aquest aminoàcid ve determinat per l'anticodó de l'ARNt. L'anticodó és una unitat composta de tres nucleòsids que corresponen a les tres bases del codó en l'ARNm. Cada ARNt conté una seqüència específica del triplet anticodó en el llaç α que pot aparellar les bases d'un o més codons per a un aminoàcid. Per exemple, el codó de la lisina és AAA, l'anticodó del corresponent ARNt seria UUU.
Visió general
Mentre que la seqüència de nucleòtids específica d’un mRNA especifica quins aminoàcids s’incorporen al producte proteic del gen del qual es transcriu l’mRNA, el paper del tRNA és el d’especificar quina seqüència del codi genètic correspon a quin aminoàcid.[1] El mRNA codifica una proteïna com una sèrie de codons contigus, cadascun dels quals és reconegut per un mRNA particular. Un dels extrems del tRNA coincideix amb el codi genètic en una seqüència de tres nucleòtids anomenada anticodó. L’anticodó forma tres parells de bases complementàries amb un codó en el tRNA durant la biosíntesi de proteïnes. A l’altre extrem del tRNA hi ha un enganxament covalent a l’aminoàcid que correspon a la seqüència anticodon. Cada tipus de molècula de tRNA es pot unir a un sol tipus d'aminoàcid, de manera que cada organisme té molts tipus d'tRNA. Com que el codi genètic conté múltiples codons que especifiquen el mateix aminoàcid, hi ha diverses molècules de tRNA amb diferents anticodons que porten el mateix aminoàcid. Durant la síntesi de proteïnes, els tRNA amb aminoàcids units s’entreguen al ribosoma mitjançant proteïnes anomenades factors d’elongació, que ajuden a l’associació del tRNA amb el ribosoma, la síntesi del nou polipèptid i la translocació (moviment) del ribosoma al llarg del mRNA. Si l'anticodó del mRNA coincideix amb el mRNA, un altre mRNA ja vinculat al ribosoma transfereix la cadena polipeptídica creixent des del seu extrem 3 a l'aminoàcid unit al 3 'extrem del recent mRNA lliurat, una reacció catalitzada pel ribosoma. Un gran nombre de nucleòtids individuals en una molècula de tRNA pot ser modificat químicament, sovint per metilació o desamidació. Aquestes bases poc freqüents afecten de vegades la interacció de el tRNA amb ribosomes i de vegades es produeixen a l’anticodó per alterar les propietats de combinació de bases.[2]
Estructura
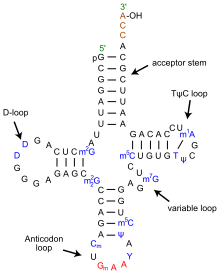
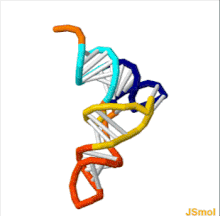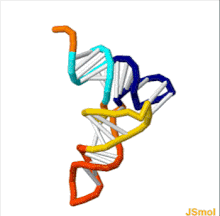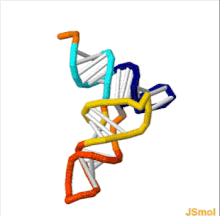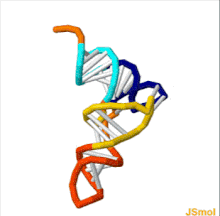
L'estructura de tRNA es pot descompondre en la seva estructura primària, la seva estructura secundària (normalment visualitzat com l'estructura de fulla de trèvol), i la seva estructura terciària[3] (tots els tRNA tenen una estructura 3D en forma de L similar que els permet encaixar-se als llocs P i A del ribosoma). L’estructura de trèvol es converteix en l’estructura en forma de L 3D mitjançant l’apilament coaxial de les helices, que és un motiu d’estructura terciària de RNA comuna. Les longituds de cada braç, així com el "diàmetre" del bucle, en una molècula de tRNA varien d'una espècie a una altra.[3][4]
Anticodó
Un anticodó[5] és una unitat formada per tres nucleòtids que corresponen a les tres bases del codó en el mRNA. Cada tRNA conté una seqüència de triplet anticodó diferent que pot formar 3 parells de bases complementàries a un o més codons per a un aminoàcid. Alguns anticodons poden combinar-se amb més d’un codó a causa d’un fenomen conegut com a aparellament de bases wobble. Sovint, el primer nucleòtid de l’anticodó no es troba a l’ARNm: la inosina, que pot unir hidrogen a més d’una base en la posició del codó corresponent.[2] En el codi genètic, és freqüent que un sol aminoàcid sigui especificat per les quatre possibilitats de tercera posició, o almenys per les pirimidines i les purines; per exemple, l'aminoàcid glicina està codificat per les seqüències de codons GGU, GGC, GGA i GGG. Altres nucleòtids modificats també poden aparèixer a la primera posició anticodon, de vegades coneguda com la "posició de balanceig", que produeixen canvis subtils al codi genètic, com per exemple en els mitocondris.[6] Per cèl·lula, es requeririen 61 tipus de tRNA per proporcionar una correspondència individual entre molècules de tRNA i codons que especifiquen aminoàcids, ja que hi ha 61 codons de sentit del codi genètic estàndard. Tanmateix, moltes cèl·lules contenen menys de 61 tipus de tRNA perquè la base ondulada és capaç de lligar-se a diversos codons que, encara que no necessàriament a tots, especifiquen un determinat aminoàcid. Cal traduir, sense ambigüitat, els 61 codons de sentit, amb un mínim de 31 tRNA; el màxim observat és de 41.[1][7]
Gens del tRNA
Els organismes varien en el nombre de gens de tRNA al seu genoma. Per exemple, el cuc del nematode C. elegans, un organisme model utilitzat habitualment en estudis de genètica, té 29.647[8] gens del seu genoma cel·lular, dels quals 620 codifiquen el tRNA.[9][10] El llevat en infància Saccharomyces cerevisiae té 275 gens de ARNt en el seu genoma.
Al genoma humà, que segons les estimacions de gener de 2013, té uns 20.848 gens codificants de proteïnes[11] en total, hi ha 497 nuclis cel·lulars que codifiquen molècules de tRNA citoplasmàtiques i pseudògens derivats de 324 mRNA -els gens del tRNA que es creu que ja no són funcionals[12] (tot i que s'ha demostrat que el pseudo-tRNA estan involucrats en la resistència als antibiòtics en bacteris).[13] També s’han identificat regions en nuclis de cromosomes, molt similars en seqüència als gens de tRNA mitocondrials.[14] Aquests aspectes també es consideren part del nucli del DNA mitocondrial (gens transferits del mitocondri al nucli).[14][15]
Com en tots els eucariotes, hi ha 22 gens de tRNA mitocondrials[16] en humans. Les mutacions d'alguns d'aquests gens s'han associat a malalties greus com la síndrome MELAS.
Els gens del tRNA citoplasmàtics es poden agrupar en 49 famílies segons les seves característiques anticodòniques. Aquests gens es troben a tots els cromosomes, excepte el cromosoma 22 i Y. S'observa un gran agrupament en 6p (gens de 140 mRNA), així com en un 1 cromosoma.[12]
L’HGNC, en col·laboració amb la base de dades de mRNA genòmic(GtRNAdb) i experts en la matèria, han aprovat noms únics per a gens humans que codifiquen els tRNA.
Referències
- ↑ 1,0 1,1 «The origin of the genetic code». Journal of Molecular Biology, vol. 38, 3, December 1968, pàg. 367–79. DOI: 10.1016/0022-2836(68)90392-6. PMID: 4887876.
- ↑ 2,0 2,1 Biochemistry. 5th. San Francisco: W.H. Freeman, 2002. ISBN 978-0-7167-4955-4.
- ↑ 3,0 3,1 «Tertiary structure of bacterial selenocysteine tRNA». Nucleic Acids Research, vol. 41, 13, July 2013, pàg. 6729–38. DOI: 10.1093/nar/gkt321. PMC: 3711452. PMID: 23649835.
- ↑ «Diversity of tRNA genes in eukaryotes». Nucleic Acids Research, vol. 34, 21, 29-10-2006, pàg. 6137–46. DOI: 10.1093/nar/gkl725. PMC: 1693877. PMID: 17088292.
- ↑ «Use of thermal denaturation studies to investigate the base sequence of yeast serine sRNA». Proceedings of the National Academy of Sciences of the United States of America, vol. 51, 5, May 1964, pàg. 818–26. Bibcode: 1964PNAS...51..818F. DOI: 10.1073/pnas.51.5.818. PMC: 300168. PMID: 14172997.
- ↑ «A complete landscape of post-transcriptional modifications in mammalian mitochondrial tRNAs». Nucleic Acids Research, vol. 42, 11, June 2014, pàg. 7346–57. DOI: 10.1093/nar/gku390. PMC: 4066797. PMID: 24831542.
- ↑ Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J. (2004). Molecular Biology of the Cell. WH Freeman: New York. 5th ed.Plantilla:ISBN?
- ↑ WormBase web site, http://www.wormbase.org, release WS187, date 25-Jan-2008.
- ↑ «Overview of gene structure». WormBook, January 2006, pàg. 1–10. DOI: 10.1895/wormbook.1.65.1. PMC: 4781370. PMID: 18023127.
- ↑ Hartwell LH, Hood L, Goldberg ML, Reynolds AE, Silver LM, Veres RC. (2004). Genetics: From Genes to Genomes 2nd ed. McGraw-Hill: New York. p. 264.
- ↑ Ensembl release 70 - Jan 2013 http://www.ensembl.org/Homo_sapiens/Info/StatsTable?db=core
- ↑ 12,0 12,1 «Initial sequencing and analysis of the human genome». Nature, vol. 409, 6822, February 2001, pàg. 860–921. Bibcode: 2001Natur.409..860L. DOI: 10.1038/35057062. PMID: 11237011.
- ↑ «A pseudo-tRNA modulates antibiotic resistance in Bacillus cereus». PLOS ONE, vol. 7, 7, 2012, pàg. e41248. Bibcode: 2012PLoSO...741248R. DOI: 10.1371/journal.pone.0041248. PMC: 3399842. PMID: 22815980.
- ↑ 14,0 14,1 «Nuclear and mitochondrial tRNA-lookalikes in the human genome». Frontiers in Genetics, vol. 5, 2014, pàg. 344. DOI: 10.3389/fgene.2014.00344. PMC: 4189335. PMID: 25339973.
- ↑ «Nuclear insertions of mitochondrial origin: Database updating and usefulness in cancer studies». Mitochondrion, vol. 11, 6, November 2011, pàg. 946–53. DOI: 10.1016/j.mito.2011.08.009. PMID: 21907832.
- ↑ Ibid. p. 529.

