Petit ARN llaç
|
|
Aquest article o secció necessita millorar una traducció deficient. |
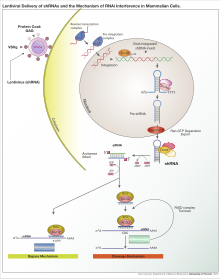
En l'àmbit de la biotecnologia, un petit ARN llaç o shRNA (de l'anglès small hairpin RNA) és una molècula d'ARN artificial amb una estructura en forma de llaç que pot ser utilitzada per reprimir l'expressió d'un gen objectiu mitjançant interferència d'ARN (RNAi, de l'anglès RNA interference).[1][2] L'expressió d'shRNA en les cèl·lules s'assoleix típicament mitjançant l'administració de plasmidis o vectors virals o bacterians. Els shRNA tenen l'avantatge de ser bons mediadors de la interferència perquè tenen una degradació lenta. Tanmateix, el seu ús requereix un vector d'expressió, fet que planteja preocupacions sobre la seguretat d'aquest mètode.[3]
L'elecció del promotor és essencial per aconseguir una expressió elevada de shRNA. Al principi, els promotors de la polimerasa d'ARN III com ara U6 i H1 van ser utilitzats; però, malauradament, aquests promotors no tenen control temporal ni espacial.[3] Per aquesta raó, hi ha hagut un tendència de canvi cap a utilitzar promotors polimerasa d'ARN II per regular millor l'expressió de shRNAs.
Administració[modifica]
L'ús d'un vector bacterià per obtenir expressió d'shRNA en les cèl·lules és una mètode relativament recent. Es basa en un estudi científic en què, mitjançant la ingestió per ratolins d'Escherichia coli recombinant amb un plasmidi que expressava shRNA, es va aconseguir reprimir l'expressió d'un gen objectiu en l'epiteli intestinal.[4] Aquest mètode va ser utilitzat l'any 2012 en assaigs clínics per provar de tractar pacients amb poliposi adenomatosa familiar.[5]
L'expressió de shRNA en les cèl·lules pot ser aconseguida mitjançant l'administració de plasmidis o a través de vectors virals o bacterians. L'administració de plasmidis a les cèl·lules via transfecció per obtenir expressió d'shRNA pot ser aconseguida in vitro utilitzant reactius disponibles comercialment. Tanmateix, aquest mètode no és aplicable in vivo i per tant té una utilitat limitada.
Diversos vectors virals poden ser utilitzats per obtenir expressió de shRNA en les cèl·lules, incloent-hi virus adenoassociats (AAVs, de l'anglès adeno-associated viruses), adenovirus i lentivirus. Amb virus adenoassociats i adenovirus, els genomes romanen com a episomes. Això és avantatjós perquè s'evita la mutagènesi per inserció, però és desavantatjós perquè la descendència de la cèl·lula perdrà el virus de pressa a través de la divisió cel·lular llevat que la cèl·lula es divideixi molt a poc a poc. Els AAV difereixen dels adenovirus en el fet que els gens virals han estat eliminats i la capacitat d'empaquetament ha estat disminuïda. Els lentivirus s'integren a seccions de cromatina amb activitat transcripcional elevada i degut a això són transmesos en la descendència de les cèl·lules. Amb aquest mètode hi ha un risc augmentat de mutagènesi per inserció; tanmateix, el risc pot ser reduït utilitzant un lentivirus deficient en integrasa, la proteïna encarregada de d'afegir el genoma viral al genoma cel·lular.[6]
Mecanisme d'acció[modifica]

Una vegada que el vector s'ha integrat en el genoma de l'hoste, l'shRNA és transcrit en el nucli per l'RNA polimerasa II o III, depenent de l'elecció del promotor. El producte imita l'estructura de llaç de pre-microARN i és processat per l'enzim Drosha. El pre-shRNA resultant és exportat del nucli per l'acció de la proteïna exportina 5. Aquest producte és llavors processat pel Dicer i després s'uneix al complex silenciador induït per ARN (RISC, de l'anglès RNA-induced silencing complex). La cadena sentit (passatgera) és degradada. La cadena antisentit (guia) dirigeix el RISC a l'ARNm que tingui una seqüència complementària. En el cas de complementarietat perfecta, el RISC escindeix l'ARNm. En el cas de complementarietat imperfecta, el RISC reprimeix la traducció de l'ARNm. En ambdós casos, la presència de l'shRNA comporta repressió gènica.
Aplicacions en teràpia gènica[modifica]
La teràpia basada en shRNA encara presenta bastants reptes. El més significatiu, com passa amb altres mètodes de teràpia gènica, és el mecanisme d'administració. L'shRNA és típicament administrat a través d'un vector, i tot i que és un mètode generalment eficaç, planteja dubtes importants sobre la seva seguretat. En particular, s'ha demostrat en estudis clínics que els mètodes de teràpia gènica basats en virus són perillosos. En la primera generació de teràpia gènica retroviral, alguns pacients de la síndrome de Wiskott-Aldrich que van ser tractats amb vectors virals van desenvolupar leucèmia aguda de limfòcits T, fet que va ser atribuït a la inserció del vector.[8] La possible sobresaturació del complex molecular RISC és també un problema. Si l'shRNA és expressat a nivells massa alts, la cèl·lula podria no ser capaç de processar correctament l'ARN endogen, fet que podria causar problemes importants. Un altre repte és la possibilitat que el pacient podria desenvolupar una resposta immunitària contra la teràpia.[9] Finalment, encara que és difícil, l'shRNA podria silenciar altre gens de manera no intencionada. Per al desenvolupament amb èxit d'un nou tipus de teràpia gènica basada en shRNA, tots d'aquests reptes s'han de tenir en compte.
Tot i aquests reptes, a causa de l'habilitat del shRNA per proporcionar repressió gènica altament específica i duradora, hi ha hagut un gran interès per utilitzar shRNA en aplicacions de teràpia gènica. Tres exemples de teràpies basades en shRNA són:
- L'empresa Gradalis va desenvolupar la vacuna FANG (ullal en anglès), que es fa servir en el tractament de càncers avançats. FANG es basa en un shRNA bifuncional (bi-shRNA) contra els factors de creixement immunosupressors transformants β1 i β2.[10] Les cèl·lules tumorals autòlogues van ser collides de pacients i un plasmidi que codifica el bi-shRNA i el factor estimulador de colònies de granulòcits i macròfags (GM-CSF) va ser introduït ex vivo mitjançant electroporació. Aquestes cèl·lules van ser més tard injectades al pacient.
- Marina Biotech va desenvolupar el CEQ508, que es fa servir per tractar la poliposi adenomatosa familiar. El CEQ508 utilitza un vector bacterià per administrar shRNA en contra de la β-catenina.
- Gradalis va desenvolupar el bi-shRNA-STMN1 (pbi-shRNA STMN1), que es fa servir per a tractar càncers avançats o metastàtics. Aquest shRNA actua contra l'estatmina 1 (oncoproteïna 18) i és administrat intratumoralment mitjançant un mètode anomenat lipoplexe de vesícules bilamel·lars invaginades.
Vegeu també[modifica]
Referències[modifica]
- ↑ Paddison, PJ; Caudy, AA; Bernstein, E; Hannon, GJ; Conklin, DS «Short hairpin RNAs (shRNAs) induce sequence-specific silencing in mammalian cells.». Genes & Development, 16, 8, 15-04-2002, pàg. 948–58. DOI: 10.1101/gad.981002. PMID: 11959843.
- ↑ Brummelkamp, TR; Bernards, R; Agami, R «A system for stable expression of short interfering RNAs in mammalian cells.». Science, 296, 5567, 19-04-2002, pàg. 550–3. DOI: 10.1126/science.1068999. PMID: 11910072.
- ↑ 3,0 3,1 Wang, Zhaohui; Rao, Donald D.; Senzer, Neil; Nemunaitis, John «RNA Interference and Cancer Therapy». Pharmaceutical Research, 28, 12, 2011, pàg. 2983–2995. DOI: 10.1007/s11095-011-0604-5. ISSN: 0724-8741.
- ↑ Xiang, Shuanglin; Fruehauf, Johannes; Li, Chiang J «Short hairpin RNA–expressing bacteria elicit RNA interference in mammals». Nature Biotechnology, 24, 6, 2006, pàg. 697–702. DOI: 10.1038/nbt1211. ISSN: 1087-0156.
- ↑ Burnett, John C.; Rossi, John J.; Tiemann, Katrin «Current progress of siRNA/shRNA therapeutics in clinical trials». Biotechnology Journal, 6, 9, 2011, pàg. 1130–1146. DOI: 10.1002/biot.201100054. ISSN: 1860-6768.
- ↑ Lombardo, Angelo; Genovese, Pietro; Beausejour, Christian M; Colleoni, Silvia; Lee, Ya-Li; Kim, Kenneth A; Ando, Dale; Urnov, Fyodor D; Galli, Cesare «Gene editing in human stem cells using zinc finger nucleases and integrase-defective lentiviral vector delivery». Nature Biotechnology, 25, 11, 2007, pàg. 1298–1306. DOI: 10.1038/nbt1353. ISSN: 1087-0156.
- ↑ Macrae I, Zhou K, Li F, Repic A, Brooks A, Cande W, Adams P, Doudna J «Structural basis for double-stranded RNA processing by dicer». Science, 311, 5758, 2006, pàg. 195–8. DOI: 10.1126/science.1121638. PMID: 16410517.
- ↑ «Solving the Problem of γ-Retroviral Vectors Containing Long Terminal Repeats». Molecular Therapy, 19, 2, 2011, pàg. 229–231. DOI: 10.1038/mt.2010.305.
- ↑ «Silencing or stimulation? siRNA delivery and the immune system». Annu Rev Chem Biomol Eng, 2, 2011, pàg. 77–96. DOI: 10.1146/annurev-chembioeng-061010-114133. PMID: 22432611.
- ↑ Senzer, Neil; Barve, Minal; Kuhn, Joseph; Melnyk, Anton; Beitsch, Peter; Lazar, Martin; Lifshitz, Samuel; Magee, Mitchell; Oh, Jonathan «Phase I Trial of "bi-shRNAifurin/GMCSF DNA/Autologous Tumor Cell" Vaccine (FANG) in Advanced Cancer». Molecular Therapy, 20, 3, 2011, pàg. 679–686. DOI: 10.1038/mt.2011.269. ISSN: 1525-0016. PMC: 3293620. PMID: 22186789.
