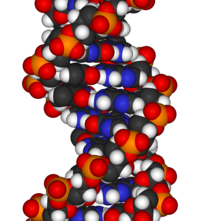
Modelització molecular de l'ADN
Els models moleculars de les estructures de l'ADN són representacions de la topologia i la geometria molecular de l'àcid desoxiribonucleic (ADN) fent ús d'un dels diversos mètodes que existeixen per a fer-ho. Tenen com a principal objectiu simplificar i presentar les propietats essencials, tant físiques com químiques, de les estructures moleculars de l'àcid desoxiribonucleic en la seva forma in vivo i in vitro. Aquestes representacions consisteixen en esferes condensades fetes de plàstic (models de CPK), filferros metàl·lics per a l'esquelet, càlculs gràfics i animacions artístiques fetes mitjançant un ordinador. A més, aquests models informatitzats de l'ADN permeten fer representacions animades i simulacions dinàmiques de la seva estructura que són de gran importància per a la comprensió de com treballa in vivo la molècula d'ADN.
Els models moleculars de l'ADN més innovadors fets a partir de programes informàtics inclouen simulacions de les seves dinàmiques moleculars, així com càlculs de la mecànica quàntica dels moviments vibracionals i rotacionals, dels orbitals moleculars deslocalitzats (abreviatura anglesa: MOs), dels moments dipolars elèctrics i dels ponts d'hidrogen, entre d'altres. En aquestes representacions també s'hi poden trobar simulacions de la geometria molecular de l'ADN i canvis en la seva topologia produïts al llarg del temps causats per les interaccions tant intra com intermoleculars d'aquesta molècula. Mentre que els models moleculars de l'àcid desoxiribonucleic (ADN) com els consistents en esferes condensades (models CPK) fetes de plàstic i filferro pels braços de l'esquelet són útils per a les representacions estàtiques de l'estructura de la molècula, tenen poca funcionalitat per a les representacions complexes de les dinàmiques de l'ADN. Els models informatitzats de la molècula permeten animacions i simulacions dinàmiques que, com s'ha expressat anteriorment, tenen una gran importància per a l'estudi del funcionament de l'ADN in vivo.
Història
[modifica]
Des de les etapes més primàries de l'estudi de les estructures de la molècula d'ADN mitjançant l'ús de difracció de raigs X i de mètodes bioquímics, els models moleculars tals com el model de la doble hèlix de Watson-Crick van ser exitosament utilitzades per a resoldre el trencaclosques que representava l'estructura de l'ADN, i també per descriure quina relació tenia aquesta amb les funcions claus de la molècula en les cèl·lules vives. Els patrons del primer sistema refractor de rajos X de qualitat foren descrits per Rosalind Franklin i Raymond Gosling, l'any 1953.[1] Els primers càlculs sobre la transformada de Fourier d'una hèlix atòmica van ser fets un any abans per Cochran, Crick i Vand,[2] i l'any 1953 foren seguits pels càlculs sobre la transformada de Fourier del model bobina enroscada pel mateix Crick.[3]
La informació estructural és generada a partir d'estudis de la difracción de rajos X sobre fibres d'ADN orientades, amb l'ajuda de models moleculars d'ADN combinats amb anàlisis cristal·logràfics i matemàtics dels patrons de rajos X.
Els primers informes que parlaven del model molecular en forma de dobre hèlix de l'estructura del B-ADN van ser efectuats per Watson i Crick l'any 1953.[4][5] Finalment, Maurice F. Wilkins, A. Stokes i H. R. Wilson van decriure els primers patrons de rajos X de B-ADN in vivo en caps d'espermatozous de salmó parcialment orientats.[6] El desenvolupament del primer model molecular de l'ADN en forma de doble hèlix correcte fet per Crick i Watson no hauria estat possible sense les evidències bioquímiques sobre l'aparellament de bases nitrogenades ([A – T] i [C – G]), o les regles de Chargaff.[7][8][9][10][11][12]
Tot i que els estudis inicials de les estructures d'ADN amb l'ajuda de models moleculars ho representaven d'una manera estàtica, les seves conseqüències en l'explicació de les funcions in vivo de l'ADN van ser molt significants en àrees com la biosíntesi de proteïnes o la universalitat del codi genètic. Tanmateix, els estudis de transformació epigenètica d'ADN in vivo van ser molt més lentes de desenvolupar tot i la seva importància de cara a l'embriologia, la morfogènesi o la recerca en el càncer. Aquestes dinàmiques de reaccions químiques i bioquímiques de l'ADN són molt més complexes que les dinàmiques moleculars de les interaccions físiques de l'ADN amb l'aigua, els ions i proteïnes i enzims en les cèl·lules vives.
Importància
[modifica]Per a més informació: Estructura dels àcids nucleics
Un dels problemes sobre dinàmica més antics, i que roman permanent i difícil de resoldre amb el pas del temps, és com té lloc el mecanisme d'autorreplicació de l'ADN en les cèl·lules vives, el qual ha d'involucrar el desenrotllament temporal de les fibres d'ADN superenrotllat. Encara que l'ADN consisteixi amb molècules de biopolímers relativament rígides i molt allargades, anomenades fibres o cadenes, formades per la repetició de quatre unitats bàsiques de nucleòtids unides a desoxiriboses i a grups fosfats; l'estructura molecular de l'ADN in vivo es sotmet a canvis dinàmics de configuració que impliquen l'aparició de molècules d'aigua i ions units dinàmicament. A més, l'assemblatge, el superenrotllament amb histones en les estructures cromosòmiques i altres aspectes supramoleculars també impliquen la topologia de l'ADN in vivo que és encara molt més complexa que la geometria molecular de l'ADN. Per tant, d'aquesta manera, el modelatge molecular de l'ADN es torna en un problema especialment difícil tant pels biòlegs moleculars com pels biotecnòlegs. Igual que altres molècules llargues i biopolímers, l'ADN normalment existeix en múltiples geometries estables, el que significa que presenta isomeria conformacional i, a més, l'ADN també existeix en múltiples estats quàntics configuracionals propers entre ells pel que fa a l'energia de la superfície d'energia potencial de la molècula d'ADN. Les diferents geometries moleculars també es poden calcular, almenys en un principi, mitjançant l'ús de mètodes ab initio de la química quàntica els quals poden assolir una alta precisió per petites molècules. Tot i així, s'han fet recents reclamacions en la base de dades de l'espectre VCD on s'afirma que una acceptable precisió també pot ser assolida tant per a polinucleòtids com per conformacions d'ADN. Per altra banda, les geometries quàntiques defineixen una classe important de models moleculars d'ADN ab initio, l'exploració dels quals tot just ha començat, especialment en relació amb els resultats obtinguts per VCD en solucions. En principi, es poden obtenir comparacions més detallades amb aquest tipus de computacions quàntiques ab initio a través de l'espectroscòpia de 2D-FT RMN i també a través d'estudis sobre la relaxació de solucions de polinuclèotids o ADN etiquetats específicament, com per exemple els ADN etiquetats amb deuteri. En un interessant gir dels papers, la molècula d'ADN s'ha proposat per ser utilitzada per a la computació quàntica. D'aquesta manera, s'han construït tant nanoestructures d'ADN com bioxips de computació d'ADN.
Conceptes fonamentals
[modifica]L'estructura química de l'ADN és insuficient per a entendre la complexitat de les estructures tridimensionals de les mateixes molècules d'ADN. Tot i així, per altra banda, els models moleculars animats de l'ADN permeten a un explorar visualment la seva l'estructura tridimensional. Així, el model d'ADN que es mostra a la dreta és un model de boles o un model CPK (“closely packed spheres”, esferes estretament empaquetades), el model de la doble hèlix de l'ADN. Aquest model és un dels que permet aquesta visualització de l'estructura tridimensional. Tot i així, hi ha altres models moleculars animats com el de tipus cable o el de tipus esquelet que es mostren al principi de l'article, que també permeten explorar visualment aquesta estructura tridimensional de la molècula d'ADN. La dinàmica dels ponts d'hidrogen i de l'intercanvi de protons és molt diferent en diversos ordres de magnitud pel que fa a dos sistemes; el sistema de l'ADN completament hidratat i el de les molècules d'aigua en gel. Per tant, la dinàmica de l'ADN és complexa, implicant nanosegons i diverses desenes en escales de temps dels picosegons. Així, la dinàmica de l'ADN en gel líquid es du a terme en l'escala de temps de picosegons mentre que l'intercanvi de protons en gel líquid es troba en l'escala de temps dels mil·lisegons. A més, les tarifes d'intercanvi de protons en l'ADN i en proteïnes unides a aquest pot variar des de picosegons a nanosegons, minuts o anys, depenent de la ubicació exacta dels protons intercanviats en els biopolímers grans. La vibració d'un oscil·lador harmònic simple és només una representació simplificada de la dinàmica de les vibracions longitudinals de les hèlixs d'ADN entrellaçades, les quals es va trobar que eren anharmòniques en contra d'harmòniques, el que s'havia assumit sovint en les simulacions dinàmiques quàntiques de l'ADN.
Estructura de l'ADN
[modifica]L'estructura de l'ADN mostra una varietat de formes, tant de cadena simple com de cadena doble. Les propietats mecàniques de l'ADN, que estan relacionades directament amb la seva estructura, són un problema significant per les cèl·lules. Cada procés que uneix o llegeix l'ADN és capaç d'utilitzar o modificar les propietats mecàniques de l'ADN per propòsits de reconeixement, empaquetatge i modificació. La longitud extrema (un cromosoma pot contenir una cadena d'ADN 10 cm de llargària), la rigidesa relativa i l'estructura helicoidal de l'ADN han portat a l'evolució de les histones i d'enzims com les topoisomerases i helicases per manejar l'ADN d'una cèl·lula. Les propietats de l'ADN estan estretament relacionades amb la seva seqüència i estructura molecular, particularment la feblesa dels enllaços d'hidrogen i les interaccions electròniques que mantenen les cadenes d'ADN juntes en comparació amb la força dels enllaços entre cada cadena. Tècniques experimentals que poden mesurar directament les propietats mecàniques de l'ADN són relativament noves i la visualització d'alta resolució en solucions és sovint difícil. No obstant això, els científics han descobert una gran quantitat de dades sobre les propietats mecàniques d'aquest polímer, per això les implicacions de les propietats mecàniques de l'ADN en processos cel·lulars és actualment un tema d'investigació actiu. L'ADN trobat en moltes cèl·lules pot ser de longitud macroscòpica - uns pocs centímetres de longitud per cada cromosoma humà. Conseqüentment, les cèl·lules han de compactar o empaquetar l'ADN per portar-lo al seu interior. Així, als eucariotes l'ADN es condensa amb proteïnes de tipus rodet conegudes com a histones, al voltant de les quals s'enrotlla l'ADN. És el màxim grau de compactació d'aquest complex ADN-proteïna que produeix els coneguts cromosomes mitòtics eucariotes.
Determinació de l'estructura de l'ADN utilitzant modelatge molecular i patrons de raigs X
[modifica]Després d'haver separat i purificat l'ADN a partir de tècniques bioquímiques estàndards, podem obtenir una mostra en un recipient (exactament com la imatge que tenim al començament de l'article). A la imatge del costat, tal com s'indica, tenim els passos a seguir per tal d'obtenir informació estructural a partir dels estudis de difracció de raigs X que es poden fer en les fibres d'ADN orientades que s'obtenen a partir de les mostres d'ADN hidratatamb l'ajuda dels models d'ADN, combinats amb anàlisis cristal·logràfics i matemàtics dels patrons de difracció dels raigs X.
Models estructurals de xarxa paracristal·lina de B-ADN
[modifica]
Una xarxa paracristal·lina és un model estructural, tant molecular com atòmic, que inclou una gran quantitat d'enllaços de desordre parcial entre els elements que la componen (un percentatge significant). Malgrat això, hi ha certs casos, com pot ser el de les nanoestructures (p. ex. vidres, o líquids), que presenten una conformació similar, però només s'hi pot observar un cert ordre local a algunes zones. Un exemple senzill de l'ordre global que implica una xarxa cristal·lina, juntament amb els seus enllaços o punts de desordre és la de la xarxa de sílice (o SiO2, com veiem a la imatge del costat). Els cristalls líquids també tenen característiques més pròpies de les xarxes paracristal·lines que de les estructures cristal·lines. Les cadenes altament hidratades de B-ADN solen aparèixer naturalment en les cèl·lules presentant aquesta estructura, ja que és molt més dinàmica que la doble hèlix helicoidal d'ADN estabilitzada per ponts d'hidrogen entre les bases complementàries dels nucleòtids de les cadenes antiparal·leles, que és relativament més rígida. Per simplicitat, la majoria dels models moleculars d'ADN ometen tant els ions com les molècules d'aigua que, de forma també dinàmica, s'uneixen al B-ADN, i són, en conseqüència, molt menys útils per entendre el comportament in vivo de les cadenes de B-ADN. L'ànalisi física i matemàtica que podem fer gràcies als raigs X[13][14] i l'espectroscòpia ens permeten veure que la xarxa paracristal·lina de B-ADN és molt més complicada que la cristal·lina que podem observar en els patrons de difracció de l'A-ADN. El model paracristal·lí és també molt important a novell tecnològic, ja que ha permès avenços com la nanotecnologia de l'ADN. Les tècniques més avançades, que estan essent desenvolupades en l'actualitat, combinaran difracció a partir de rajos X i microscòpia electrònica en cèl·lules hidratades i vives.[15]
Aplicacions genòmiques i biotecnològiques del modelatge molecular de l'ADN
[modifica]
El modelatge molecular de l'ADN té diverses aplicacions en les investigacions de Genòmica i Biotecnologia, des de la reparació de l'ADN fins a la PCR (Reacció en Cadena de la Polimerasa, anomenada així per les sigles en anglès “Polymerase Chain Reaction”) o les nanoestructures d'ADN. A més, ja s'ha aconseguit veure imatges del model bidimensional en qüestió (com veiem a la imatge de dalt, amb microscopis de forces atòmiques[16]), que presenta l'aspecte d'una xarxa molt endreçada. Com ja s'ha comentat, el modelatge molecular té moltes aplicacions, i en diferents àrees. Entre aquestes, també trobem la computació de models moleculars de molècules tan diverses i variades com la RNA polimerasa, la plantilla de la primasa de l'ADN bacteri (que suggereix una dinàmica molt complexa en les interfícies entre enzims-DNA), i també models de la interacció química i mutàgena entre les molècules carcinògenes amb l'ADN de les cèl·lules. A la galeria que s'adjunta a continuació es veuen imatges del models esmentats. Entre les seves nombroses aplicacions també contem els bioxips d'ADN, les nanoestructures d'ADN dissenyades per la computació de l'ADN i altres aplicacions dinàmiques de la nanotecnologia de l'ADN.[17][18][19][20][21][22] A la representació anterior podem veure estructures fruit de l'autoacoblament de molècules d'ADN. La xarxa d'ADN que s'hi observa permet veure una estructura que consisteix en quatre ramificacions orientades a 90° que fan d'unió entre les molècules. Cada ramificació conté nou oligonucleòtids d'ADN (com veiem a la figura de la dreta de la imatge que encapçala l'apartat). Aquestes ramificacions venen a ser el “bloc constitutiu” primari de l'assemblatge de les molècules, que culmina amb l'estructura que veiem a la figura de l'esquerra. El DNA quàdruplex pot estar involucrat en alguns càncers[23][24]. Veiem imatges que exemplifiquen i il·lustren aquest tipus d'estructures en la galeria que s'adjunta a continuació.
Galeria d'imatges
[modifica]-
Model de boles genèric d'ADN giratori.
-
Dibuix que mostra un model ultra-simplificat de l'estructura de doble hèlix de l'ADN.
-
Model de replicació de l'ADN basat en el concepte de la doble hèlix.
-
Model molecular animat de boles de la doble hèlix del A-ADN.
-
Model de Watson i Crick d'ADN det a gran escala (Museo Príncipe Felipe, Valencia).
-
Vista lateral del models moleculars dels tres tipus d'ADN (A, B i Z).
-
Model ultra-simplificat de la doble hèlix d'A-ADN.
-
Modelatge molecular de la ARN Polimerasa.
-
Modelatge molecular d'una plantilla de Primasa del DNa bacterià.
-
Modelatge de les reparacions moleculars de l'ADN amb la molècula MGMT.
-
Model molecular tridimensional del dany de l'ADN causat pel carcinogen 2-AF.
-
Modelatge molecular de la reparació d'ADN.
-
Model esquemàtic animat de l'A-ADN.
-
Models simplificats de cromatina.
-
Model simplificat de l'estructura d'un coromosoma.
-
Model de l'ADN quàdruplex hipotètic ric en estructures de guaninaque podrien tenir a veure amb el càncer.
-
Estructura molecular tridimensional del telòmer d'una estructura quadrúplexa en solució de potassi (visió intramolecular).
-
Model molecular de boles de l'ADN.
-
Model d'un tetraedre d'ADN dissenyat.
Vegeu també ...
[modifica]- G-quàdruplex
- Cristal·lografia
- Estructura cristal·lina
- Microscopi de raigs X
- Difracció de raigs X
- Difracció de neutrons
- Dicroïsme vibracional rotacional (VCD)
- Espectroscòpia Raman/microscòpia i CARS
- Sir Lawrence Bragg
- Llista de software de simulació d'àcids nucleics
- AMBER
- CHARMM
- Abalone (mecàniques moleculars)
- Software de visualització Sírius
- Espectroscòpia de ressonància magnètica nuclear
- Imatgeria per ressonància magnètica
- Espectroscòpia per microones
- Espectroscòpia per infrarojos
- Espectroscòpia per “infrarojos propers”
- Tècniques d'obtenció d'imatges per espectre, per hiperespectre, i amb química
- Correlació espectroscòpica fluorescent
- Espectroscòpia de correlació transversal fluorescent i FRET
- Microscòpia confocal
Referències
[modifica]- ↑ Franklin, R.E., Gosling, R.G. (6 March 1953). "The Structure of Sodium Thymonucleate Fibres I. The Influence of Water Content". Acta Cryst. 6: 673. doi:10.1107/S0365110X53001939. Franklin, R.E., Gosling, R.G. (6 March 1953). "The Structure of Sodium Thymonucleate Fibres II. The Cylindrically Symmetrical Patterson Function". Acta Cryst. 6: 678. doi:10.1107/S0365110X53001940.
- ↑ Cochran, W., Crick, F.H.C., Vand V. (1952). "The Structure of Synthetic Polypeptides. 1. The Transform of Atoms on a Helix". Acta Cryst. 5 (5): 581–6. doi:10.1107/S0365110X52001635.
- ↑ Crick, F.H.C. (1953a). "The Fourier Transform of a Coiled-Coil". Acta Crystallographica 6 (8-9): 685–9. doi:10.1107/S0365110X53001952.
- ↑ Watson, James D., Crick, Francis H.C. (25 April 1953). "A structure for Deoxyribose Nucleic Acid" (PDF). Nature 171: 737–8. doi:10.1038/171737a0. http://www.nature.com/nature/dna50/watsoncrick.pdf
- ↑ Watson, J.D; Crick F.H.C. (1953b). "The Structure of DNA". Cold Spring Harbor Symposia on Quantitative Biology 18: 123–31. PMID: 13168976
- ↑ Wilkins M.H.F., A.R. Stokes A.R. & Wilson, H.R. (1953). "Molecular Structure of Deoxypentose Nucleic Acids" (PDF). Nature 171 (4356): 738–40. doi:10.1038/171738a0. PMID: 13054693. http://www.nature.com/nature/dna50/wilkins.pdf
- ↑ Elson D, Chargaff E (1952). "On the deoxyribonucleic acid content of sea urchin gametes". Experientia 8 (4): 143–5. doi:10.1007/BF02170221. PMID: 14945441
- ↑ Chargaff E, Lipshitz R, Green C (1952). "Composition of the deoxypentose nucleic acids of four genera of sea-urchin". J Biol Chem 195 (1): 155–160. PMID: 14938364
- ↑ Chargaff E, Lipshitz R, Green C, Hodes ME (1951). "The composition of the deoxyribonucleic acid of salmon sperm". J Biol Chem 192 (1): 223–230. PMID: 14917668
- ↑ Chargaff E (1951). "Some recent studies on the composition and structure of nucleic acids". J Cell Physiol Suppl 38 (Suppl)
- ↑ Magasanik B, Vischer E, Doniger R, Elson D, Chargaff E (1950). "The separation and estimation of ribonucleotides in minute quantities". J Biol Chem 186 (1): 37–50. PMID: 14778802
- ↑ Chargaff E (1950). "Chemical specificity of nucleic acids and mechanism of their enzymatic degradation". Experientia 6 (6): 201–9. doi:10.1007/BF02173653. PMID: 15421335
- ↑ Hosemann R., Bagchi R.N., Direct analysis of diffraction by matter, North-Holland Publs., Amsterdam – New York, 1962.
- ↑ Baianu, I.C. (1978). "X-ray scattering by partially disordered membrane systems.". Acta Cryst., A34 (5): 751–3. doi:10.1107/S0567739478001540.
- ↑ Yamamoto Y, Shinohara K (October 2002). "Application of X-ray microscopy in analysis of living hydrated cells". Anat. Rec. 269 (5): 217–23. doi:10.1002/ar.10166. PMID: 12379938
- ↑ Mao, Chengde; Sun, Weiqiong & Seeman, Nadrian C. (16 June 1999). "Designed Two-Dimensional DNA Holliday Junction Arrays Visualized by Atomic Force Microscopy". Journal of the American Chemical Society 121 (23): 5437–43. doi:10.1021/ja9900398
- ↑ Robinson, Bruche H.; Seeman, Nadrian C. (August 1987). "The Design of a Biochip: A Self-Assembling Molecular-Scale Memory Device". Protein Engineering 1 (4): 295–300. ISSN 0269-2139
- ↑ Rothemund, Paul W. K.; Ekani-Nkodo, Axel; Papadakis, Nick; Kumar, Ashish; Fygenson, Deborah Kuchnir & Winfree, Erik (22 December 2004). "Design and Characterization of Programmable DNA Nanotubes". Journal of the American Chemical Society 126 (50): 16344–52. doi:10.1021/ja044319l. PMID: 15600335
- ↑ Keren, K.; Kinneret Keren, Rotem S. Berman, Evgeny Buchstab, Uri Sivan, Erez Braun (November 2003). "DNA-Templated Carbon Nanotube Field-Effect Transistor". Science 302 (6549): 1380–2. doi:10.1126/science.1091022. PMID: 14631035. http://www.sciencemag.org/cgi/content/abstract/sci;302/5649/1380
- ↑ Zheng, Jiwen; Constantinou, Pamela E.; Micheel, Christine; Alivisatos, A. Paul; Kiehl, Richard A. & Seeman Nadrian C. (2006). "2D Nanoparticle Arrays Show the Organizational Power of Robust DNA Motifs". Nano Letters 6 (7): 1502–4. doi:10.1021/nl060994c. PMID: 16834438
- ↑ Cohen, Justin D.; Sadowski, John P.; Dervan, Peter B. (2007). "Addressing Single Molecules on DNA Nanostructures". Angewandte Chemie 46 (42): 7956–9. doi:10.1002/anie.200702767. PMID: 17763481
- ↑ Constantinou, Pamela E.; Wang, Tong; Kopatsch, Jens; Israel, Lisa B.; Zhang, Xiaoping; Ding, Baoquan; Sherman, William B.; Wang, Xing; Zheng, Jianping; Sha, Ruojie & Seeman, Nadrian C. (2006). "Double cohesion in structural DNA nanotechnology". Organic and Biomolecular Chemistry 4 (18): 3414–9. doi:10.1039/b605212f. PMID: 17036134
Bibliografia
[modifica]- Applications of Novel Techniques to Health Foods, Medical and Agricultural Biotechnology.(June 2004) I. C. Baianu, P. R. Lozano, V. I. Prisecaru and H. C. Lin., q-bio/0406047.
- F. Bessel, Untersuchung des Theils der planetarischen Störungen, Berlin Abhandlungen (1824), article 14.
- Sir Lawrence Bragg, FRS. The Crystalline State, A General survey. London: G. Bells and Sons, Ltd., vols. 1 and 2., 1966., 2024 pages.
- Cantor, C. R. and Schimmel, P.R. Biophysical Chemistry, Parts I and II., San Franscisco: W.H. Freeman and Co. 1980. 1,800 pages.
- Voet, D. and J.G. Voet. Biochemistry, 2nd Edn., New York, Toronto, Singapore: John Wiley & Sons, Inc., 1995, ISBN 0-471-58651-X., 1361 pages.
- Watson, G. N. A Treatise on the Theory of Bessel Functions., (1995) Cambridge University Press. ISBN 0-521-48391-3.
- Watson, James D. Molecular Biology of the Gene. New York and Amsterdam: W.A. Benjamin, Inc. 1965., 494 pages.
- Wentworth, W.E. Physical Chemistry. A short course., Malden (Mass.): Blackwell Science, Inc. 2000.
- Herbert R. Wilson, FRS. Diffraction of X-rays by proteins, Nucleic Acids and Viruses., London: Edward Arnold (Publishers) Ltd. 1966.
- Kurt Wuthrich. NMR of Proteins and Nucleic Acids., New York, Brisbane,Chicester, Toronto, Singapore: J. Wiley & Sons. 1986., 292 pages.
- Hallin PF, David Ussery D (2004). "CBS Genome Atlas Database: A dynamic storage for bioinformatic results and DNA sequence data". Bioinformatics 20 (18): 3682–6. doi:10.1093/bioinformatics/bth423. PMID: 15256401.
- Zhang CT, Zhang R, Ou HY (2003). "The Z curve database: a graphic representation of genome sequences". Bioinformatics 19 (5): 593-599. doi:10.1093/bioinformatics/btg041