Glicoproteïna d'espícula (coronavius)
 | |
| Substància | família de proteïnes |
|---|---|
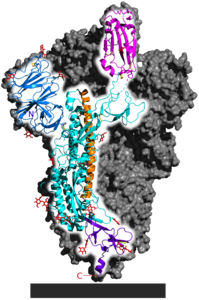
La glicoproteïna[1] d'espícula[2] del SARS-CoV-2 és una estructura proteica que es projecta cap a l'exterior (peplòmer) del virió SARS-CoV-2. És una glicoproteïna transmembrana de la superfície del virus del SARS-CoV-2 i serveix com a lligand per acoblar-se a l'ACE2 a la superfície cel·lular i com a proteïna fusogènica per a l'entrada cel·lular. Igual que amb els altres membres dels Coronaviridae (del llatí: Corona), les glicoproteïnes d'espícula formen el distintiu i homònim de l'estructura superficial d'aquests virions.
Característiques[modifica]


El virió SARS-CoV-2 utilitza les glicoproteïnes d'espícula per unir-se al receptor ACE2 de les cèl·lules corporals (cèl·lules hostes), amb les quals es fusiona i es multiplica.[4]
Unió i fusió amb les cèl·lules hostes[modifica]
La glicoproteïna d'espícula és una proteïna homotrimèrica i una de les quatre proteïnes estructurals del SARS-CoV-2.[5][6] Determina l'hoste i la cèl·lula Tropisme del SARS-CoV-2.[6] [7] Hi ha entre 15 i 30 homotrímers de la glicoproteïna d'espícula que poden girar lliurement a la superfície d'un virió.[8] Cadascun dels tres monòmers idèntics té tres ponts disulfur interns.[5] Cadascuna de les tres subunitats iguals es divideix en dues parts S1 i S2 mitjançant proteòlisi per una proteasa semblant a la furina abans de la posició 686, amb les parts escindides S1 i S2 romanent lligades entre si. A diferència d'altres coronavirus, la interfície té la seqüència R R A R, que facilita la divisió.[9] A l'àrea S1, després d'un pèptid senyal, hi ha primer el domini N -terminal (NTD), seguit del domini d'unió al receptor (RBD) per acoblar-se a la cèl·lula hoste.[10] El pèptid senyal conté 13 aminoàcids que estan disposats en espiral i de manera hidròfoba (que evita l'aigua).[10] El NTD conté la seqüència GTNGTKR, que presumiblement permet la unió a altres proteïnes de membrana.[8] La N-glicosilació a N 165 i N234 estabilitza la unió del RBD a ACE2.[3] La N -glicosilació a N61 i N603 condueix a una major accessibilitat de la interfície per a les proteases.[8]
Després de l'absorció a l' endosoma, S2 és escindit encara més per TMPRSS2 abans de la posició 816, creant S2' (posició 816 - 1273) i activant el domini de fusió.[7] [11] Aquesta escissió no està recolzada essencialment per la catepsina L. [12] A l'àrea S2 es troben el pèptid de fusió (FP) i el pèptid de fusió interna (IFP) per a la fusió del virus i la membrana cel·lular, dos dominis de repetició d'hèptades (HR1 i HR2), un domini transmembrana i finalment un domini C -terminal curt. es troba al citosol.[10] A S2', falta el pèptid de fusió FP a causa de l'escissió.[10] Les proteïnes SARS-CoV-2 M i E modulen el transport de proteïnes de glicoproteïnes d'espícula de nova formació mitjançant diferents mecanismes.[13] El domini transmembrana és la segona seqüència d'aminoàcids hidrofòbics de la glicoproteïna d'espícula.[10]
Plecs proteics[modifica]

La glicoproteïna d'espícula té tres possibles plecs de proteïnes : la forma de prefusió nativa, la forma de bucle de forquilla i la forma de bucle de forquilla de postfusió. Durant la fusió de la membrana amb la cèl·lula hoste, les estructures de bobines enrotllades (amb patró heptade ) formen un trímer de bucles de forquilla, apropant el pèptid de fusió a la regió C -terminal del domini proteic extracel·lular, després de la qual es fusiona la membrana amb la membrana de la cèl·lula hoste. es produeix.[5] La unió al receptor indueix un canvi en el plegament que fa que el pèptid de fusió estigui més exposat, provocant així la fusió de la membrana.[16] El pèptid de fusió consta de dues parts a les posicions 816 – 837 i 835 – 855.[5] El domini transmembrana es troba als aminoàcids 1214 – 1236.[5] A través de la fusió, l'interior del virus (anglès: core ) que conté el genoma viral s'allibera al citosol, després de la qual cosa comença la traducció de les proteïnes virals.[6] Com moltes altres proteïnes fusogèniques, les glicoproteïnes d'espícula dels coronavirus humans [17] i també la del SARS-CoV-2 condueixen a la formació de sincitia,[18][19] presumiblement a la infecció de cèl·lules veïnes.[10]
Algunes vacunes contra el SARS-CoV-2 utilitzen una variant de la glicoproteïna S com a antigen, que té dues prolines modificades a prop del domini de fusió que estabilitzen el plegament de proteïnes abans de la fusió de la membrana (estabilitzat per prefusió 2P). A la variant 2P, es van intercanviar dos aminoàcids per prolina: a la posició 1060 abans hi havia una valina, a la posició 1061 abans hi havia una leucina.[20] La variant 2P es va descriure per primera vegada per als coronavirus a MERS-CoV.[20] Es va confirmar l'analogia de la variant 2P en SARS-CoV-2.[21]
immunologia[modifica]


Els anticossos contra la glicoproteïna d'espícula es formen després de la infecció per SARS-CoV-2, així com la vacunació amb vacunes COVID-19. Els anticossos neutralitzants contra el SARS-CoV-2 s'uneixen en una proporció d'uns 90 % al RBD a S1.[16] [11] Els anticossos monoclonals bamlanivimab, casirivimab, cilgavimab, etesevimab, imdevimab, regdanvimab, sotrovimab i tixagevimab s'uneixen al domini d'unió al receptor (RBD) de la glicoproteïna d'espícula del SARS-CoV-2. Molts epítops a la superfície de la glicoproteïna d'espícula estan emmascarats per glicosilacions extensives.[3] Com que el RBD està menys emmascarat per glicosilacions en comparació amb altres estructures superficials, el RBD és presumiblement immunodominant per neutralitzar els anticossos.[22] Les mutacions d'escapament d'anticossos es produeixen principalment en una regió específica del RBD, que també indica la immunodominància del RBD per als anticossos.[23] Els anticossos neutralitzants contra la glicoproteïna d'espícula es divideixen en 4 classes segons les seves propietats d'unió: [24]
- Anticossos que bloquegen l'ACE2 que s'uneixen a la forma de plec obert de la glicoproteïna d'espícula (classe 1)
- Anticossos que bloquegen l'ACE2 que s'uneixen als plecs oberts i tancats de la glicoproteïna d'espícula (classe 2)
- Anticossos que no bloquegen l'ACE2 que s'uneixen als plecs oberts i tancats del RBD de la glicoproteïna d'espícula (classe 3)
- Anticossos neutralitzants que s'uneixen fora del lloc d'unió de l'ACE2 i només al plec obert (classe 4)
Els pocs anticossos neutralitzants que no s'uneixen al RBD s'uneixen majoritàriament al NTD.[16] S'uneixen amb freqüència a les posicions 14-20, 140-158 i 245-264.[25]
Hi ha reactivitat creuada d'anticossos neutralitzants contra la glicoproteïna S en ratolins, inhibint tant l'entrada de cèl·lules SARS-CoV com SARS-CoV-2.[26] Els dos coronavirus associats al SARS (1 i 2) utilitzen el mateix receptor per a l'entrada cel·lular, l'enzim convertidor d'angiotensina 2 (ACE2), mentre que MERS-CoV utilitza dipeptidil peptidasa 4 (CD26).[27] La superfície d'unió a ACE2 difereix entre el RBD de les glicoproteïnes d'espícula de SARS-CoV-1 i -2, per això no tots els seus anticossos neutralitzants són de reacció creuada.[10] S1 és més variable que S2 en la seqüència d'aminoàcids, per això és més probable que els anticossos d'unió a S2 tinguin reacció creuada.[10]
A la glicoproteïna d'espícula del SARS-CoV-2, es van identificar 13 epítops per a MHC I (generen una resposta immune cel·lular) i 3 per a MHC II (generen una resposta immune humoral) per als humans.[28] S'han identificat epítops conservats en la glicoproteïna S i la proteïna nucleocàpsida que poden ser adequades per a vacunes àmpliament efectives.[29]
Mutacions[modifica]
Les mutacions a la glicoproteïna d'espícula del SARS-CoV-2 sorgeixen durant la replicació del genoma viral. Es tracta principalment de mutacions amb costos de fitness nuls o baixos.[16] A més, també s'observen mutacions que tenen un fort impacte en la seqüència d'aminoàcids de la glicoproteïna d'espícula.[16] Aquests solen afectar la patogenicitat, la infecciositat, la transmissibilitat i/o l'antigenicitat.[16] Algunes de les mutacions de major impacte han sorgit diverses vegades i de manera independent en la seqüència de glicoproteïnes d'espícula al llarg de la pandèmia de Covid-19.[16]
A la posició 614, del domini d'unió al receptor (RBD), les mutacions no sinònimes es produeixen amb més freqüència, cosa que indica una selecció positiva més forta per a aquest lloc a la glicoproteïna d'espícula.[16] Les mutacions D614G, N501Y, E484K-Q, K417N/T i L452R s'associen amb un augment de la producció de virus, una major transmissibilitat, una virulència alterada i probablement també amb una fugida immune.[30][31] La mutació D614 G confereix una major infectivitat i transmissibilitat del SARS-CoV-2.[16] [32][33] La mutació N 439 K condueix a una major afinitat pel receptor i redueix la unió d'alguns anticossos neutralitzants.[16] La mutació Y 453 F també condueix a una major afinitat per ACE2.[16] Les mutacions a la posició 484 condueixen a una unió reduïda mitjançant anticossos neutralitzants i, per tant, són mutacions d'escapament,[34] especialment E 484K, E484 Q, E484 P, E484 A, E484D i E484G.[16] [35] També a la posició les mutacions K444R, K444 N, K444Q, V445E i a la posició 477 les mutacions S 477G, S477N i S477R.[16] K417N/T, L452R, Y453F, S477N i N501Y s'han descrit com a mutacions d'escapament d'anticossos addicionals en altres posicions.[36][37][33]
A la NTD, les regions 140–156 i 246–260 s'han descrit com a llocs per a mutacions d'escapament, incloent N148S, K150R, K150E, K150T, K150Q i S151P.[16] La supressió dels aminoàcids a les posicions 69 i 70, a la NTD, augmenta la infectivitat.[16] A la variant omicron, les mutacions 478K, E484A, Q493R i N501Y tenen la influència més forta entre els seus més de 30 aminoàcids canviats a la glicoproteïna d'espícula.[38]
Ús[modifica]
En vacunes[modifica]
Totes les vacunes aprovades contenen la glicoproteïna d'espícula o el seu gen com a antigen. Sovint s'utilitza la variant 2P, en què el plegament de proteïnes es va estabilitzar abans que el virus es fusionés amb la membrana cel·lular. Això pretén "reduir el risc teòric de generar anticossos que milloren la infecció".[39]
En assaigs de neutralització[modifica]
Per examinar les mostres de sang per determinar la seva capacitat per neutralitzar el SARS-CoV-2, a causa del perill del virus, només s'utilitzen virus pseudotipatsen lloc dels virions reals; aquests són virus modificats ("quimèrics") que contenen la glicoproteïna d'espícula de Conté SARS-CoV-2.[40][41] S'utilitzen vectors lentivirals que també tenen proteïnes d'espícula a la superfície, amb el codi genètic de les glicoproteïnes d'espícula (lentivirus) substituït pel codi de l'espiga SARS-CoV-2.
Referències[modifica]
- ↑ «Optimot. Consultes lingüístiques». [Consulta: 22 setembre 2023].
- ↑ «Optimot. Consultes lingüístiques». [Consulta: 22 setembre 2023].
- ↑ 3,0 3,1 3,2 L. Casalino, Z. Gaieb, J. A. Goldsmith, C. K. Hjorth, A. C. Dommer, A. M. Harbison, C. A. Fogarty, E. P. Barros, B. C. Taylor, J. S. McLellan, E. Fadda, R. E. Amaro: Beyond Shielding: The Roles of Glycans in the SARS-CoV-2 Spike Protein. In: ACS central science. Band 6, Nummer 10, Oktober 2020, S. 1722–1734, doi:10.1021/acscentsci.0c01056, PMID 33140034, PMC 7523240
- ↑ Mechanisms of SARS-CoV-2 entry into cells (www.nature.com/, 5. Oktober 2021)
- ↑ 5,0 5,1 5,2 5,3 5,4 «UniProt». [Consulta: 22 setembre 2023].
- ↑ 6,0 6,1 6,2 Y. Wang, M. Grunewald, S. Perlman: Coronaviruses: An Updated Overview of Their Replication and Pathogenesis. In: Methods in molecular biology. Band 2203, 2020, S. 1–29, doi:10.1007/978-1-0716-0900-2_1, PMID 32833200, PMC 7682345
- ↑ 7,0 7,1 C. Zhu, G. He, Q. Yin, L. Zeng, X. Ye, Y. Shi, W. Xu: Molecular biology of the SARs-CoV-2 spike protein: A review of current knowledge. In: Journal of medical virology. Band 93, Nummer 10, 10 2021, S. 5729–5741, doi:10.1002/jmv.27132, PMID 34125455, PMC 8427004
- ↑ 8,0 8,1 8,2 S. B. Kadam, G. S. Sukhramani, P. Bishnoi, A. A. Pable, V. T. Barvkar: SARS-CoV-2, the pandemic coronavirus: Molecular and structural insights. In: Journal of basic microbiology. Band 61, Nummer 3, März 2021, S. 180–202, doi:10.1002/jobm.202000537, PMID 33460172, PMC 8013332
- ↑ R. Kumavath, D. Barh, B. S. Andrade, M. Imchen, F. F. Aburjaile, A. Ch, D. L. Rodrigues, S. Tiwari, K. J. Alzahrani, A. Góes-Neto, M. E. Weener, P. Ghosh, V. Azevedo: The Spike of SARS-CoV-2: Uniqueness and Applications. In: Frontiers in immunology. Band 12, 2021, S. 663912, doi:10.3389/fimmu.2021.663912, PMID 34305894, PMC 8297464
- ↑ 10,0 10,1 10,2 10,3 10,4 10,5 10,6 10,7 X. Xia: Domains and Functions of Spike Protein in Sars-Cov-2 in the Context of Vaccine Design. In: Viruses. Band 13, Nummer 1, Januar 2021, S. , doi:10.3390/v13010109, PMID 33466921, PMC 7829931
- ↑ 11,0 11,1 P. V'kovski, A. Kratzel, S. Steiner, H. Stalder, V. Thiel: Coronavirus biology and replication: implications for SARS-CoV-2. In: Nature reviews. Microbiology. Band 19, Nummer 3, 03 2021, S. 155–170, doi:10.1038/s41579-020-00468-6, PMID 33116300, PMC 7592455
- ↑ C. B. Jackson, M. Farzan, B. Chen, H. Choe: Mechanisms of SARS-CoV-2 entry into cells. In: Nature reviews. Molecular cell biology. Band 23, Nummer 1, 01 2022, S. 3–20, doi:10.1038/s41580-021-00418-x, PMID 34611326, PMC 8491763
- ↑ B. Boson, V. Legros, B. Zhou, E. Siret, C. Mathieu, F. L. Cosset, D. Lavillette, S. Denolly: The SARS-CoV-2 envelope and membrane proteins modulate maturation and retention of the spike protein, allowing assembly of virus-like particles. In: Journal of Biological Chemistry. Band 296, 2021 Jan-Jun, S. 100111, doi:10.1074/jbc.RA120.016175, PMID 33229438, PMC 7833635
- ↑ A. C. Walls, X. Xiong, Y. J. Park, M. A. Tortorici, J. Snijder, J. Quispe, E. Cameroni, R. Gopal, M. Dai, A. Lanzavecchia, M. Zambon, F. A. Rey, D. Corti, D. Veesler: Unexpected Receptor Functional Mimicry Elucidates Activation of Coronavirus Fusion. In: Cell. Band 176, Nummer 5, 02 2019, S. 1026–1039.e15, doi:10.1016/j.cell.2018.12.028, PMID 30712865, PMC 6751136
- ↑ X. Fan, D. Cao, L. Kong, X. Zhang: Cryo-EM analysis of the post-fusion structure of the SARS-CoV spike glycoprotein. In: Nature Communications. Band 11, Nummer 1, 07 2020, S. 3618, doi:10.1038/s41467-020-17371-6, PMID 32681106, PMC 7367865
- ↑ 16,00 16,01 16,02 16,03 16,04 16,05 16,06 16,07 16,08 16,09 16,10 16,11 16,12 16,13 16,14 W. T. Harvey, A. M. Carabelli, B. Jackson, R. K. Gupta, E. C. Thomson, E. M. Harrison, C. Ludden, R. Reeve, A. Rambaut,., S. J. Peacock, D. L. Rober: SARS-CoV-2 variants, spike mutations and immune escape. In: Nature reviews. Microbiology. Band 19, Nummer 7, 07 2021, S. 409–424, doi:10.1038/s41579-021-00573-0, PMID 34075212, PMC 8167834
- ↑ S. Belouzard, J. K. Millet, B. N. Licitra, G. R. Whittaker: Mechanisms of coronavirus cell entry mediated by the viral spike protein. In: Viruses. Band 4, Nummer 6, 06 2012, S. 1011–1033, doi:10.3390/v4061011, PMID 22816037, PMC 3397359
- ↑ J. Buchrieser, J. Dufloo, M. Hubert, B. Monel, D. Planas, M. M. Rajah, C. Planchais, F. Porrot, F. Guivel-Benhassine, S. Van der Werf, N. Casartelli, H. Mouquet, T. Bruel, O. Schwartz: Syncytia formation by SARS-CoV-2-infected cells. In: The EMBO Journal. Band 39, Nummer 23, 12 2020, S. e106267, doi:10.15252/embj.2020106267, PMID 33051876, PMC 7646020
- ↑ Z. Zhang, Y. Zheng, Z. Niu, B. Zhang, C. Wang, X. Yao, H. Peng, D. N. Franca, Y. Wang, Y. Zhu, Y. Su, M. Tang, X. Jiang, H. Ren, M. He, Y. Wang, L. Gao, P. Zhao, H. Shi, Z. Chen, X. Wang, M. Piacentini, X. Bian, G. Melino, L. Liu, H. Huang, Q. Sun: SARS-CoV-2 spike protein dictates syncytium-mediated lymphocyte elimination. In: Cell death and differentiation. Band 28, Nummer 9, 09 2021, S. 2765–2777, doi:10.1038/s41418-021-00782-3, PMID 33879858, PMC 8056997
- ↑ 20,0 20,1 J. Pallesen, N. Wang, K. S. Corbett, D. Wrapp, R. N. Kirchdoerfer, H. L. Turner, C. A. Cottrell, M. M. Becker, L. Wang, W. Shi, W. P. Kong, E. L. Andres, A. N. Kettenbach, M. R. Denison, J. D. Chappell, B. S. Graham, A. B. Ward, J. S. McLellan: Immunogenicity and structures of a rationally designed prefusion MERS-CoV spike antigen. In: Proceedings of the National Academy of Sciences. Band 114, Nummer 35, 08 2017, S. E7348–E7357, doi:10.1073/pnas.1707304114, PMID 28807998, PMC 5584442
- ↑ D. Wrapp, N. Wang, K. S. Corbett, J. A. Goldsmith, C. L. Hsieh, O. Abiona, B. S. Graham, J. S. McLellan: Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. In: Science. Band 367, Nummer 6483, 03 2020, S. 1260–1263, doi:10.1126/science.abb2507, PMID 32075877, PMC 7164637
- ↑ Y. Watanabe, Z. T. Berndsen, J. Raghwani, G. E. Seabright, J. D. Allen, O. G. Pybus, J. S. McLellan, I. A. Wilson, T. A. Bowden, A. B. Ward, M. Crispin: Vulnerabilities in coronavirus glycan shields despite extensive glycosylation. In: Nature Communications. Band 11, Nummer 1, 05 2020, S. 2688, doi:10.1038/s41467-020-16567-0, PMID 32461612, PMC 7253482
- ↑ A. J. Greaney, A. N. Loes, K. H. Crawford, T. N. Starr, K. D. Malone, H. Y. Chu, J. D. Bloom: Comprehensive mapping of mutations in the SARS-CoV-2 receptor-binding domain that affect recognition by polyclonal human plasma antibodies. In: Cell host & microbe. Band 29, Nummer 3, 03 2021, S. 463–476.e6, doi:10.1016/j.chom.2021.02.003, PMID 33592168, PMC 7869748
- ↑ C. O. Barnes, C. A. Jette, M. E. Abernathy, K. A. Dam, S. R. Esswein, H. B. Gristick, A. G. Malyutin, N. G. Sharaf, K. E. Huey-Tubman, Y. E. Lee, D. F. Robbiani, M. C. Nussenzweig, A. P. West, P. J. Bjorkman: SARS-CoV-2 neutralizing antibody structures inform therapeutic strategies. In: Nature. Band 588, Nummer 7839, 12 2020, S. 682–687, doi:10.1038/s41586-020-2852-1, PMID 33045718, PMC 8092461
- ↑ M. McCallum, A. De Marco, F. A. Lempp, M. A. Tortorici, D. Pinto, A. C. Walls, M. Beltramello, A. Chen, Z. Liu, F. Zatta, S. Zepeda, J. di Iulio, J. E. Bowen, M. Montiel-Ruiz, J. Zhou, L. E. Rosen, S. Bianchi, B. Guarino, C. S. Fregni, R. Abdelnabi, S. C. Foo, P. W. Rothlauf, L. M. Bloyet, F. Benigni, E. Cameroni, J. Neyts, A. Riva, G. Snell, A. Telenti, S. P. Whelan, H. W. Virgin, D. Corti, M. S. Pizzuto, D. Veesler: N-terminal domain antigenic mapping reveals a site of vulnerability for SARS-CoV-2. In: Cell. Band 184, Nummer 9, 04 2021, S. 2332–2347.e16, doi:10.1016/j.cell.2021.03.028, PMID 33761326, PMC 7962585
- ↑ A. C. Walls, Y. J. Park, M. A. Tortorici, A. Wall, A. T. McGuire, D. Veesler: Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein. In: Cell. [elektronische Veröffentlichung vor dem Druck] März 2020, doi:10.1016/j.cell.2020.02.058, PMID 32155444.
- ↑ E. Prompetchara, C. Ketloy, T. Palaga: Immune responses in COVID-19 and potential vaccines: Lessons learned from SARS and MERS epidemic. In: Asian Pacific journal of allergy and immunology. [elektronische Veröffentlichung vor dem Druck] März 2020, doi:10.12932/AP-200220-0772, PMID 32105090.
- ↑ M. Bhattacharya, A. R. Sharma, P. Patra, P. Ghosh, G. Sharma, B. C. Patra, S. S. Lee, C. Chakraborty: Development of epitope-based peptide vaccine against novel coronavirus 2019 (SARS-COV-2): Immunoinformatics approach. In: Journal of medical virology. [elektronische Veröffentlichung vor dem Druck] Februar 2020, doi:10.1002/jmv.25736, PMID 32108359.
- ↑ S. F. Ahmed, A. A. Quadeer, M. R. McKay: Preliminary Identification of Potential Vaccine Targets for the COVID-19 Coronavirus (SARS-CoV-2) Based on SARS-CoV Immunological Studies. In: Viruses. Band 12, Nummer 3, Februar 2020, S. , doi:10.3390/v12030254, PMID 32106567.
- ↑ V. Papanikolaou, A. Chrysovergis, V. Ragos, E. Tsiambas, S. Katsinis, A. Manoli, S. Papouliakos, D. Roukas, S. Mastronikolis, D. Peschos, A. Batistatou, E. Kyrodimos, N. Mastronikolis: From delta to Omicron: S1-RBD/S2 mutation/deletion equilibrium in SARS-CoV-2 defined variants. In: Gene. Band 814, März 2022, S. 146134, doi:10.1016/j.gene.2021.146134, PMID 34990799, PMC 8725615
- ↑ B. Bakhshandeh, Z. Jahanafrooz, A. Abbasi, M. B. Goli, M. Sadeghi, M. S. Mottaqi, M. Zamani: Mutations in SARS-CoV-2; Consequences in structure, function, and pathogenicity of the virus. In: Microbial Pathogenesis. Band 154, Mai 2021, S. 104831, doi:10.1016/j.micpath.2021.104831, PMID 33727169, PMC 7955574
- ↑ D. C. Groves, S. L. Rowland-Jones, A. Angyal: The D614G mutations in the SARS-CoV-2 spike protein: Implications for viral infectivity, disease severity and vaccine design. In: Biochemical and biophysical research communications. Band 538, 01 2021, S. 104–107, doi:10.1016/j.bbrc.2020.10.109, PMID 33199022, PMC 7643658
- ↑ 33,0 33,1 C. B. Jackson, L. Zhang, M. Farzan, H. Choe: Functional importance of the D614G mutation in the SARS-CoV-2 spike protein. In: Biochemical and biophysical research communications. Band 538, 01 2021, S. 108–115, doi:10.1016/j.bbrc.2020.11.026, PMID 33220921, PMC 7664360
- ↑ I. Lazarevic, V. Pravica, D. Miljanovic, M. Cupic: Immune Evasion of SARS-CoV-2 Emerging Variants: What Have We Learnt So Far? In: Viruses. Band 13, Nummer 7, 06 2021, S. , doi:10.3390/v13071192, PMID 34206453, PMC 8310325
- ↑ E. Boehm, I. Kronig, R. A. Neher, I. Eckerle, P. Vetter, L. Kaiser: Novel SARS-CoV-2 variants: the pandemics within the pandemic. In: Clinical microbiology and infection : the official publication of the European Society of Clinical Microbiology and Infectious Diseases. Band 27, Nummer 8, August 2021, S. 1109–1117, doi:10.1016/j.cmi.2021.05.022, PMID 34015535, PMC 8127517
- ↑ D. Focosi, F. Maggi: Neutralising antibody escape of SARS-CoV-2 spike protein: Risk assessment for antibody-based Covid-19 therapeutics and vaccines. In: Reviews in Medical Virology. Band 31, Nummer 6, 11 2021, S. e2231, doi:10.1002/rmv.2231, PMID 33724631, PMC 8250244
- ↑ A. Winger, T. Caspari: The Spike of Concern-The Novel Variants of SARS-CoV-2. In: Viruses. Band 13, Nummer 6, 05 2021, S. , doi:10.3390/v13061002, PMID 34071984, PMC 8229995
- ↑ S. Kannan, P. Shaik Syed Ali, A. Sheeza: Omicron (B.1.1.529) - variant of concern - molecular profile and epidemiology: a mini review. In: European review for medical and pharmacological sciences. Band 25, Nummer 24, 12 2021, S. 8019–8022, doi:10.26355/eurrev_202112_27653, PMID 34982466.
- ↑ Antikörper COVID-19-Geimpfter erkennen andere Strukturen des Spike-Proteins als Antikörper SARS-CoV-2-Infizierter (Pressemitteilung des Paul-Ehrlich-Instituts 26/2021, aktualisiert 9. Dezember 2021, www.pei.de)
- ↑ J. M. Condor Capcha, G. Lambert, D. M. Dykxhoorn, A. G. Salerno, J. M. Hare, M. A. Whitt, S. Pahwa, D. T. Jayaweera, L. A. Shehadeh: Generation of SARS-CoV-2 Spike Pseudotyped Virus for Viral Entry and Neutralization Assays: A 1-Week Protocol. In: Frontiers in cardiovascular medicine. Band 7, 2020, S. 618651, doi:10.3389/fcvm.2020.618651, PMID 33521067, PMC 7843445
- ↑ Effective screening of SARS-CoV-2 neutralizing antibodies in patient serum using lentivirus particles pseudotyped with SARS-CoV-2 spike glycoprotein (www.nature.com, 5. November 2020)
Bibliografia[modifica]
- Zunlong Ke, Joaquin Oton et al. a.: Estructures i distribucions de proteïnes espiga SARS-CoV-2 en virions intactes. A: Natura. 588, 2020, pàg. 498, doi:10.1038/s41586-020-2665-2.
- Bálint Kiss, Zoltán Kis, Bernadett Pályi, Miklós SZ Kellermayer: topografia, dinàmica de puntes i nanomecànica dels virions SARS-CoV-2 natius individuals. A: Nano Letters. 21, 2021, pàg. 2675, doi:10.1021/acs.nanolett.0c04465.
Enllaços externs[modifica]
- Angiotensin-converting enzyme 2 in Membranome database
- 3D structure of protein complex of a neurotransmitter sodium symporter Sodium-dependent neutral amino acid transporter B(0)AT1|B(0)AT1, ACE2, and SARS-CoV-2 receptor-binding domain in OPM database
