Proteïna S del SARS-CoV-2

La glicoproteïna Spike del SARS-CoV-2, coneguda també com a proteïna S, és una glicoproteïna de fusió vírica de classe I (conformant una espícula vírica) composta per 1273 aminoàcids.[1] La proteïna S ha estat la diana terapèutica clau de cara a la cura de la COVID-19, ja sigui en forma de vacuna, anticòs terapèutic o diferents tipus de diagnòstics clínics. La seva importància radica en el fet que el SARS-CoV-2 la utilitza com a clau d'entrada per accedir dins de les cèl·lules sanes de l'hoste infectat, per medi de l'enzim conversiu de l'angiotensina 2 de la membrana cel·lular. Estructuralment, està formada per dues subunitats: la subunitat S1 i la S2. La primera s'encarrega del reconeixement i unió amb el receptor, mentre que la segona és responsable de la fusió cel·lular. Les glicoproteïnes Spike s'ajunten a la superfície vírica formant trímers, cosa que acaba resultant en la curiosa aparença exterior de corona d'aquests virus.[2]
La variabilitat vírica que presenta la subfamília del Coronavirus ve donada, en part, per les diferències en la proteïna S dels diferents membres, ja que ha anat evolucionant diferentment fins a tal punt que aquesta proteïna, entre els diferents virus de la família, no difereix només en aspectes estructurals de mida, pes i llargària, sinó que també presenta diferències funcionals, com ara en les interaccions contra receptors determinats o en la resposta a detonants naturals de la fusió de membranes entre cèl·lula i virus.[2]
Estructura bioquímica[modifica]
La proteïna S és una proteïna transmembrana del tipus I, d'una mida relativament gran que pot arribar als 180-200 kDa[3] i d'una llargada aproximada de 160Å.[4] Aquesta proteïna, a part d'estar conformada per un gran nombre d'aminoàcids, es troba considerablement glicosilada (la proteïna S dels CoV pot arribar a presentar entre 21-35 llocs per N-glicosilacions). Presenta característiques estructurals, topològiques i funcionals semblants a altres proteïnes virals de fusió del tipus I i, com aquestes, regula el mecanisme d'entrada viral mitjançant una reorganització estructural, on passa d'un estat metaestable a un estat intermedi i, finalment, a un estat estable de post-fusió.[4]

La proteïna consisteix en un fragment N-terminal extracel·lular, un domini transmembrana ancorat a la membrana viral i un petit segment C-terminal intracel·lular. La proteïna normalment presenta una conformació metaestable de prefusió, i un cop el virus interactua amb la cèl·lula hoste, aquest entra en estat de fusió gràcies a una reorganització estructural extensiva de la proteïna que permet que el virus es fusioni amb la membrana de la cèl·lula hoste.[3] A més, les proteïnes es recobreixen amb uns polisacàrids per tal de camuflar-se del sistema immunitari de l'hoste durant l'entrada.[5]
La llargada total de la proteïna és de 1273 aminoàcids. Presenta un senyal peptídic localitzat al fragment N-terminal entre els aminoàcids 1-13, la subunitat S1 (residus 14-685) i una subunitat S2 (residus 686-1273).[3] En la subunitat S1, hi ha un altre domini N-terminal (residus 14-305) i el Receptor-Binding Domain o RBD. Aquest consisteix en cinc làmines-β antiparal·leles (β1, β2, β3, β4 i β7), que formen el nucli conjuntament amb petites hèlices i bucles de connexió. Després de dos petits subdominis (SD1/2), trobem dos llocs d'escisió de proteasa (S1/S2 i S2'). A la subunitat S2, hi trobem el pèptid de fusió o Fusion Peptide (FP) (residus 788-806), dues hèptades peptídiques repetitives (HR1 entre els residus 912-984 i HR2 entre els residus 1163-1213), una hèlix central (CH), un domini connector (CD), el domini transmembrana (residus 1213-1237) i un domini citosòlic (residus 1237-1273).[3]

És crucial destacar també la divergència pel que fa als gens codificants de la proteïna S entre diversos coronavirus, atès que el gen S del SARS-CoV-2 presenta una diferència considerable respecte el dels altres CoV. La glicoproteïna S presenta un 76% de similitud amb la proteïna S del SARS-CoV Urbani quant a la seqüència aminoacídica, i un 80% amb la dels SARSr-CoV ZXC21 i ZC45[6] (aquests dos últims són soques derivades de ratpenats). Ara bé, també cal destacar la similitud genòmica que presenta la proteïna S del SARS-CoV-2 amb la del virus RaTG13, amb una similitud d'un 97%.[7] Altrament, la similitud en la seqüència aminoacídica de la regió RBD del COVID-19 es només d'un 74% i 90,1%[7] amb el SARS-CoV i RaTG13, respectivament. No obstant, malgrat la diferència evident que mostra tenir la proteïna S amb la seva homòloga en el SARS-CoV, el mecanisme d'enllaçament és similar pels dos virus.[8]
Malgrat les diferències, la conservació estructural de seqüències aminoacídiques en ambdós virus porta a la hipòtesi que la regió RBM podria induir l'anomenada "reactivitat creuada" per part d'anticossos.[8] Però, actualment no existeixen moltes evidències científiques que demostrin aquest fenomen per part d'anticossos potencials i capacitats per a dur aquesta activitat.
La glicoproteïna S del SARS-CoV-2 està glicosilada densament mitjançant N-glicans heterogenis que sobresurten de la superfície trimèrica. Aquests oligosacàrids exerceixen diferents funcions: participen en el plegament de la proteïna, segurament tenen un paper clau en la defensa del virus davant el reconeixement per anticossos i afecten al "priming" realitzat per les proteases pròpies[6] (com pot ser la furina). La separació proteolítica de les subunitats S1 i S2, procés denominat a l'anglès com a "priming", atorga a les proteïnes Spike dels coronavirus la flexibilitat estructural necessària per a la reacció de fusió de membranes.[9] Per tant, la importància bioquímica que suposen les diverses glicosilacions d'aquesta proteïna és significativa de cara a la funcionalitat final d'aquesta. En el cas del virus que s'està tractant en aquest article, presenta 22 regions per protòmer per a fer N-glicosilacions (GlcNAc) i dues regions O-glicosilacions[6][10] (vegeu la taula). Si es compara això amb la proteïna S del SARS-CoV, aquest presenta 23 regions en lloc de 22. A més, 20 regions de les 22 presentades en la proteïna S del SARS-CoV-2 es troben conservades en la proteïna S del SARS-CoV. En específic, 9 dels 13 glicans de la subunitat S1 i els 9 glicans de la S2.[6] Algunes d'aquestes regions són, per exemple: N17LT, N74GT o N1194ES.[6]
| MODIFICACIÓ | RESIDUS | MODIFICACIÓ | RESIDUS | MODIFICACIÓ | RESIDUS | MODIFICACIÓ | RESIDUS | |||
|---|---|---|---|---|---|---|---|---|---|---|
| ENLLAÇ DISULFUR | 15-136 | ENLLAÇ DISULFUR | 291-301 | N-GLICOSILACIÓ | 603 | ENLLAÇ DISULFUR | 840-851 | |||
| N-GLICOSILACIÓ | 17 | O-GLICOSILACIÓ (GalNAc) | 323 | N-GLICOSILACIÓ | 616 | ENLLAÇ DISULFUR | 1032-1043 | |||
| N-GLICOSILACIÓ | 61 | O-GLICOSILACIÓ | 325 | ENLLAÇ DISULFUR | 617-649 | N-GLICOSILACIÓ | 1074 | |||
| N-GLICOSILACIÓ | 74 | N-GLICOSILACIÓ | 331 | N-GLICOSILACIÓ | 657 | ENLLAÇ DISULFUR | 1082-1126 | |||
| N-GLICOSILACIÓ | 122 | ENLLAÇ DISULFUR | 336-361 | ENLLAÇ DISULFUR | 662-671 | N-GLICOSILACIÓ | 1098 | |||
| ENLLAÇ DISULFUR | 131-166 | N-GLICOSILACIÓ | 343 | N-GLICOSILACIÓ | 709 | N-GLICOSILACIÓ | 1134 | |||
| N-GLICOSILACIÓ | 149 | ENLLAÇ DISULFUR | 379-432 | N-GLICOSILACIÓ | 717 | N-GLICOSILACIÓ | 1158 | |||
| N-GLICOSILACIÓ | 165 | ENLLAÇ DISULFUR | 391-525 | ENLLAÇ DISULFUR | 738-760 | N-GLICOSILACIÓ | 1173 | |||
| N-GLICOSILACIÓ | 234 | ENLLAÇ DISULFUR | 480-488 | ENLLAÇ DISULFUR | 743-748 | N-GLICOSILACIÓ | 1194 | |||
| N-GLICOSILACIÓ | 282 | ENLLAÇ DISULFUR | 538-590 | N-GLICOSILACIÓ | 801 |
Quant a l'estructura secundària de la proteïna, s'han detectat avui dia diferents segments regulars i estables de la cadena polipeptídica que conforma la glicoproteïna Spike del SARS-CoV-2: 57 làmines beta, 26 hèlices alfa i 9 girs.[10]
Subunitat S1[modifica]
La subunitat S1 comprèn els dominis N-terminal (NTD), Receptor-Binding Domain (RBD) i els subdominis 1 i 2.
La unió del virus al receptor de membrana de la cèl·lula hoste és el principi de la infecció, per tant, reconèixer el receptor es un punt clau en la entrada viral. El Receptor-Binding domain (RBD) de S1 s'uneix al receptor cel·lular ACE2 en la regió aminopeptidasa N, aquest presenta substitucions en residus claus que reforcen aquesta interacció. Si comparem el SARS-CoV-2 RBD amb el SARS-RBD, veiem que la inferfície d'unió del SARS-CoV-2 RBD té més residus que interactuen amb el receptor ACE2 que el SARS-RBD, concretament el primer en té 21 i el segon 17. A més, en el complex SARS-CoV-2 RBD-ACE 2 s'hi troba enterrada una superfície més gran que amb el SARS RBD.
Les mutacions tenen un paper molt important en la millora de la interacció amb l'ACE2. Per exemple, F486 en el SARS-CoV-2 en comptes de I472 en el SARS RBD, forma fortes interaccions aromàtiques amb Y83 d'ACE2; i E484 en SARS-CoV-2-CTD, en lloc de P470 en SARS RBD, forma interaccions iòniques amb l'K31, el que condueix a una major afinitat amb el receptor que el RBD del SARS-CoV.[3]

Subunitat S2[modifica]
La subunitat S2 està composta pels dominis FP, HR1, HR2, TM i CT (cua citoplasmàtica), i és responsable de la fusió viral i de l'entrada.
- FP és un segment curt, de 15 a 20 aminoàcids conservats de la família viral, format principalment per residus hidrofòbics com ara la Glicina (G) o la Alanina (A), que s'ancoren a la membrana diana quan la proteïna adopta la conformació prehairpin.
- Pel que fa als dominis HR1 i HR2, estan formats d'un heptapèptid repetitiu: HPPHCPC, on H és un residu hidrofòbic o tradicionalment voluminós, P és un residu hidrofílic o polar, i C és un altre residu carregat. Aquests dos dominis formen un feix de sis hèlices (6-HB) que és essencial per la fusió viral i per la funció d'entrada de la subunitat S2.
- L'HR1 es troba a l'extrem C d'un FP hidrofòbic, i l'HR2 el trobem a l'extrem N del domini TM.
- El domini TM posterior, ancora la proteïna S a la membrana viral i acaba en una cua citoplasmàtica (CT).[3]
Biosíntesi[modifica]
La traducció de la proteïna S, que fa ús de la maquinària de replicació de la cèl·lula hoste, comença al citosol. El precursor sense processar conté una seqüència senyal de transport al reticle endoplasmàtic rugós (RER) a l'extrem N-terminal, que dirigeix la proteïna cap a la membrana del RER perquè continuï sent traduïda. Posteriorment, la seqüència és eliminada per les peptidases del lumen. Una única seqüència senyal stop-transfer a l'extrem C-terminal, que comprèn tota l'amplada de la membrana, evita que la proteïna s'alliberi per complet al lumen i es secreti fora de la cèl·lula hoste. Cotraduccionalent, s'uneixen cadenes d'oligosacàrids riques en manoses amb enllaços N-glicosídics. Després de la síntesi, els monòmers de glicoproteïna S formen trímers, amb l'estructura típica de corona, fet que facilita el seu transport a l'aparell de Golgi. En aquest nou compartiment la majoria de les cadenes d'oligosacàrids es tornen més complexes, i també se n'afegeixen de noves amb enllaços O-glicosídics. Al trans Golgi network (TGN) es produeix la proteòlisi al lloc de trencament S1/S2 per la furina o proteases similars, de manera que es descobreix una nova superfície a la subunitat S1 encarregada de la unió amb el receptor de membrana de la cèl·lula hoste i una altra a S2 amb la funció de fusió de les membranes del virus i l'hoste. Ambdues subunitats romanen associades mitjançant interaccions no covalents en un estat metaestable de pre-fusió. A continuació, una seqüència senyal de retorn al reticle endoplasmàtic (ERRS) assegura que la proteïna S madura s'acumula, conjuntament amb la proteïna de membrana del virus (M), al compartiment intermedi RE-Golgi, on participen per muntar el virió i s'incorporen a la membrana d'aquest. Per altra banda, una part de les proteïnes S madures viatgen per la via secretora cap a la membrana plasmàtica, on poden produir la fusió de cèl·lules infectades amb cèl·lules sanes, formant cèl·lules gegants multinucleades.[4]
Interacció amb el receptor[modifica]
El SARS-CoV2 pot infectar els pneumòcits tipus II dels alvèols mitjançant la interacció amb el receptor de l'ACE2.
La interacció es dona entre el domini d'unió del receptor (RBD) i el domini N-terminal peptidasa de l'ECA2, que està format per dos lòbuls. El contacte es produeix a la part inferior del lòbul petit de l'ACE2, amb una superfície còncava exterior del RBD, permetent l'acomodament de l'hèlix alfa N-terminal de l'ACE2 i, conseqüentment, creant una interfície de 1687 Å^2. Hi participen vàries interaccions, incloses les hidrofíliques amb 13 ponts d'hidrogen i 2 ponts salins.[3]
| Interacció | SARS-CoV-2 RBD | Distància (Å) | ECA2 |
|---|---|---|---|
| Ponts d'hidrogen | N487(ND2) | 2.6 | Q24(OE1) |
| K417(NZ) | 3.0 | D30(OD2) | |
| Q493(NE2) | 2.8 | E35(OE2) | |
| Y505(OH) | 3.2 | E37(OE2) | |
| Y449(OH) | 2.7 | D38(OD2) | |
| T500(OG1) | 2.6 | Y41(OH) | |
| N501(N) | 3.7 | Y41(OH) | |
| G446(O) | 3.3 | Q42(NE2) | |
| Y449(OH) | 3.0 | Q42(NE2) | |
| Y489(OH) | 3.5 | Y83(OH) | |
| N487(OD1) | 2.7 | Y83(OH) | |
| G502(N) | 2.8 | K353(O) | |
| Y505(OH) | 3.7 | R393(NH2) | |
| Ponts salins | K417(NZ) | 3.9 | D30(OD1) |
| K417(NZ) | 3.0 | D30(OD2) |
No obstant, la hidrofobicitat de la superfície de la proteïna S del SARS-CoV-2, major que en el cas del SARS-CoV, és el principal factor que estabilitza la unió amb el receptor, fet que podria explicar la seva alta infectivitat. Abans de l'acoblament, les cadenes laterals hidrofíliques d'ambdues proteïnes es troben fent ponts d'hidrogen amb les molècules d'aigua del medi extracel·lular. Així doncs, s'han de desfer d'aquesta capa d'aigua per tal d'unir-se entre elles. Les interaccions hidrofòbiques a la zona d'unió entre la proteïna S i l'ECA2 faciliten aquesta tasca, de manera que les cadenes laterals hidrofíliques poden participar en la interacció hidrofòbica amb les seves porcions hodrofòbiques. A la superfície del RBD es troben diversos residus amb cadenes laterals hidrofòbiques, com la glicina (Gly), alanina (Ala), valina (Val), leucina (Leu), isoleucina (Ile), prolina (Pro), fenilalanina (Phe), metionina (Met) i triptòfan (Trp).[11]
Fusió[modifica]
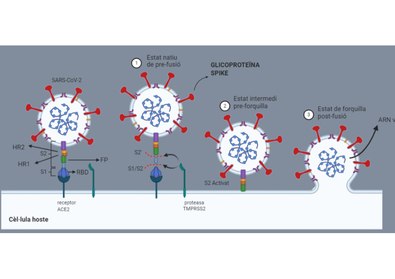
El mecanisme d'infecció comença amb la unió entre la subunitat S1 de la proteïna S i el receptor ACE2 de la cèl·lula hoste, de manera que el virió queda adherit a la membrana cel·lular. La subunitat S2 lidera la fusió del virió i les membranes cel·lulars atès a que actua com una proteïna viral de fusió de classe l. La subunitat de fusió (S2) conté una gran varietat de motius, entre ells el pèptid de fusió (FP). El FP descriu una seqüència curta (15-25 aminoàcids) composta majoritàriament per residus hidrofòbics com la glicina (G) o l'alanina (A), les quals s'insereixen en la membrana de l'hoste per desencadenar la fusió.[12] Els dominis HR1 i HR2 també són clau en la mediació d'aquests processos. Malgrat el fet que aquest procés encara està en estudi, s'ha establert un model provisional que estableix que la glicoproteïna S passa per tres estats conformacionals: estat natiu de pre-fusió, estat intermedi "pre-forquilla" (pre-hairpin) i estat de "forquilla" post-fusió.[4]
La unió del RBD de S1 amb l'enzim ACE2 produeix la partició activadora de la proteïna S en la zona que connecta S1 i S2 (prèviament units per interaccions no covalents). En conseqüència, s'exposa un punt de trencament a S2 (S2’) susceptible de ser atacat per les proteases de la cèl·lula hoste, iniciant el procés de fusió.[1] La proteòlisi a S2’, que pot ser catalitzada per una serina proteasa cel·lular (TMPRSS2) o una cisteïna proteasa endosòmica (catepsina B i L), allibera el pèptid de fusió i desestabilitza l'estructura de pre-fusió de la proteïna S. A continuació el FP s'insereix a la membrana de l'hoste, formant l'estat pre-hairpin, que a causa de la seva inestabilitat passa ràpidament a l'estat de post-fusió quan S2 es torna a doblegar de manera irreversible.[4] Aquest canvi conformacional, anomenat fusion core (nucli de fusió) o six-helix bundle (paquet de 6 hèlix), estira de les membranes del virus i l'hoste fins que es troben a una distància molt propera. El fusion core és produït pel muntatge de tres regions de HR1 en forma d'un trímer coiled-coil (hèlix superenrotllada) que s'uneix a tres regions de HR2 mitjançant interaccions hidrofòbiques.
El procés de fusió es realitza en dues etapes: l'hemifusió i la formació del porus d'entrada. L'hemifusió consisteix en la unió de les monocapes exteriors del virus i l'hoste, de manera que els continguts interiors es mantenen intactes. Finalment, es forma un porus d'entrada que permet la fusió completa entre les bicapes lipídiques i l'abocament de l'ARN víric dins la cèl·lula hoste.[12]

Usos terapèutics[modifica]
Atès a la gran importància bioquímica que aquesta proteïna suposa funcionalment per a la infectivitat i patogenicitat del virus, ha esdevingut una de les principals dianes terapèutiques dels tractaments en experimentació que s'estan estudiant i valorant actualment com a cura de la malaltia. Com que aquesta molècula és responsable de l'enllaçament amb el receptor hoste i de la fusió de membranes, la futura vacuna basada en aquesta proteïna induiria anticossos que reconeixerien i bloquejarien l'enllaçament i fusió viral i, en conseqüència, neutralitzaria la infecció vírica.
Per a desenvolupar la nova vacuna, s'estan utilitzant coneixements obtinguts a partir d'experiències anteriors amb altres virus de la família, com el SARS-CoV o el MERS-CoV. Com ja s'ha esmentat anteriorment, aquests dos virus comparteixen amb el SARS-CoV-2 certes seqüències conservades i estructures similars, motiu pel qual s'estan considerant els mecanismes d'immunització contra aquests dos virus com a guia de cara al disseny final del prototip de vacuna.[13] Les principals aproximacions al disseny de la vacuna final es fonamenten en la immunització contra un o més antígens presents en l'estructura vírica del SARS-CoV-2, ja sigui mitjançant administració directa de la proteïna purificada o per expressió d'un vector víric de vacuna (ADN o ARN).
Les proteïnes superficials del virus són les dianes principals que s'estan estudiant. A part de la proteïna S, el virus presenta també una petita proteïna E (envelope), la proteïna de matriu M i la proteïna nucleocàpside N. Estudis amb vectors recombinants que expressaven la proteïna S del SARS-CoV van trobar que aquesta molècula és altament immunogènica, mentre que les proteïnes N, M i E no ho eren tant i de fet quasi no contribuïen gens a la neutralització de la resposta per part de l'anticòs.[13] En altres estudis, s'ha vist que anticossos dirigits contra la proteïna S del SARS-CoV resultaven eficaços en relació a la inhibició de l'entrada vírica a les cèl·lules sanes. Encara més recentment, s'ha trobat que la glicoproteïna S del SARS-CoV induïa l'aparició de respostes immunes mediades per anticossos policlonals, que alhora neutralitzaven l'entrada cel·lular mediada per la proteïna S del virus SARS-CoV-2.[13] Aquest últim descobriment aporta una raó empírica significativa més que possibilita la idea que una vacuna o immunoteràpia focalitzada contra aquesta diana terapèutica podria ser efectiva.
En concret, cal destacar les estructures RBD i HR com a dianes claus de l'estructura molecular de la glicoproteïna.[3][13] La regió RBD és un objectiu crític pels anticossos neutralitzants (nAbs). Degut a la relativa similitud que presenten SARS-CoV i SARS-CoV-2 en aquestes estructures, s'ha vist experimentalment que anticossos específics contra RBD del SARS-CoV podien induir reactivitat creuada contra la regió RBD del SARS-CoV-2, trobant que l'ús de seroteràpia mitjançant antisèrum contra RBD del SARS-CoV neutralitzava el SARS-CoV-2. Aquesta és una informació important més que aporta evidències què apuntar contra aquest domini amb una vacuna podria resultar eficaç de cara a la prevenció del COVID-19.[13] Quant al domini HR, també ha mostrat ser un domini molt atractiu i interessant de cara a l'estudi de la teràpia, doncs la regió HR de la subunitat S2 té un paper vital en la infectivitat dels HCoV, a més d'estar ben conservada entre els diferents virus de la família en relació a l'estructura i a la funcionalitat (interacció entre subdominis HR1 i HR2).[3]
Tots aquests detalls i particularitats sobre la glicoproteïna (importància fonamental en la capacitat infectiva del virus, estructura extracel·lular superficial, interacció amb ACE2, conservació entre diferents coronavirus...) han fet que esdevingui el principal focus d'atenció pel que fa al desenvolupament d'anticossos neutralitzants i de qualsevol altre tipus de teràpia o vacuna.[13] Algunes de les companyies que ja han començat a estudiar aquesta possibilitat són les següents:[13]
- Altimmune: teràpia basada en un vector d'adenovirus incapaç de replicació i que incorpora la proteïna S del SARS-CoV-2, administrat per una única dosi intranasal.
- CanSino Biologicals: teràpia basada en un vector d'adenovirus del tipus 5 que expressa la proteïna S.
- Inovio Pharmaceuticals: la teràpia es fonamenta, en aquest cas, en una electroporació del DNA INO-4800 que codifica la proteïna S del SARS-CoV-2.
- Moderna: aquesta empresa basa la seva teràpia en un RNAm que codifica la proteïna S del SARS-CoV-2 encapsulat dins d'un lípid ionitzable, diestearoil fosfatidilcolina, colesterol i glicol de polietilè.
- Novavax: teràpia que consisteix en una nanopartícula que exhibeix la proteïna S del SARS-CoV-2, conjuntament amb l'aplicació d'un adjuvant immunològic basat en saponina (Matriu-M). Un adjuvant és qualsevol substància que s'afegeix a una vacuna per tal de potenciar i dirigir la teràpia contra l'antígen en concret.
No obstant això, tot i estudiar-se la possibilitat d'utilitzar aquesta proteïna com a diana terapèutica, s'està analitzant també el possible risc que podria derivar d'una hipotètica immunoteràpia focalitzada contra aquesta proteïna, al considerar la possibilitat que, degut a aquestes immunoteràpies, la COVID-19 esdevingui encara pitjor. Aquest succés hipotètic, que potenciaria i milloraria encara més el SARS-CoV-2, es deuria a un fenomen entre l'anticòs i el virus conegut com a millora depenent d'anticossos o ADE (Antibody-Dependent Enhancement). Aquest fenomen consisteix en el fet que la unió entre el virus i el seu respectiu anticòs, no produiria la neutralització o eliminació del patògen, sinó que de fet, seria un potenciador de la infecció, és a dir, milloraria i facilitaria l'entrada del virus dins de les cèl·lules. En efecte, s'ha suggerit que el motiu pel qual hi ha hagut tantes diferències geogràfiques quant a la severitat de la malaltia, és degut a l'ADE, donades les exposicions anteriors de les regions geogràfiques determinades a epítops antigènics similars. En el cas del SARS-CoV-2, l'entrada viral per ADE a la cèl·lula hoste és mediada pel receptor Fc II i no pel receptor regular.[13]
Tot i la controvèrsia que aquest últim punt està generant, experiments amb models animals de vacunes basades en la proteïna S del SARS-CoV-2 estan resultant prometedors.[13]
Vegeu també[modifica]
Referències[modifica]
- ↑ 1,0 1,1 Pillay, Tahir S. «Gene of the month: the 2019-nCoV/SARS-CoV-2 novel coronavirus spike protein» (en anglès). Journal of Clinical Pathology, 73, 7, 01-07-2020, pàg. 366–369. DOI: 10.1136/jclinpath-2020-206658. ISSN: 0021-9746. PMID: 32376714.
- ↑ 2,0 2,1 «Spike Protein / S Protein». [Consulta: 28 octubre 2020].
- ↑ 3,00 3,01 3,02 3,03 3,04 3,05 3,06 3,07 3,08 3,09 Huang, Yuan; Yang, Chan; Xu, Xin-feng; Xu, Wei; Liu, Shu-wen «Structural and functional properties of SARS-CoV-2 spike protein: potential antivirus drug development for COVID-19» (en anglès). Acta Pharmacologica Sinica, 41, 9, 2020-09, pàg. 1141–1149. DOI: 10.1038/s41401-020-0485-4. ISSN: 1745-7254.
- ↑ 4,0 4,1 4,2 4,3 4,4 Duan, Liangwei; Zheng, Qianqian; Zhang, Hongxia; Niu, Yuna; Lou, Yunwei «The SARS-CoV-2 Spike Glycoprotein Biosynthesis, Structure, Function, and Antigenicity: Implications for the Design of Spike-Based Vaccine Immunogens». Frontiers in Immunology, 11, 07-10-2020. DOI: 10.3389/fimmu.2020.576622. ISSN: 1664-3224. PMC: 7575906. PMID: 33117378.
- ↑ Watanabe, Yasunori; Allen, Joel D.; Wrapp, Daniel; McLellan, Jason S.; Crispin, Max «Site-specific glycan analysis of the SARS-CoV-2 spike». Science (New York, N.Y.), 369, 6501, 07 17, 2020, pàg. 330–333. DOI: 10.1126/science.abb9983. ISSN: 1095-9203. PMC: 7199903. PMID: 32366695.
- ↑ 6,0 6,1 6,2 6,3 6,4 «Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein» (en anglès). Cell, 181, 2, 16-04-2020, pàg. 281–292.e6. DOI: 10.1016/j.cell.2020.02.058. ISSN: 0092-8674.
- ↑ 7,0 7,1 Ou, Xiuyuan; Liu, Yan; Lei, Xiaobo; Li, Pei; Mi, Dan «Characterization of spike glycoprotein of SARS-CoV-2 on virus entry and its immune cross-reactivity with SARS-CoV» (en anglès). Nature Communications, 11, 1, 27-03-2020, pàg. 1620. DOI: 10.1038/s41467-020-15562-9. ISSN: 2041-1723.
- ↑ 8,0 8,1 Yi, Chunyan; Sun, Xiaoyu; Ye, Jing; Ding, Longfei; Liu, Meiqin «Key residues of the receptor binding motif in the spike protein of SARS-CoV-2 that interact with ACE2 and neutralizing antibodies» (en anglès). Cellular & Molecular Immunology, 17, 6, 2020-06, pàg. 621–630. DOI: 10.1038/s41423-020-0458-z. ISSN: 2042-0226.
- ↑ Hoffmann, Markus; Hofmann-Winkler, Heike; Pöhlmann, Stefan «Priming Time: How Cellular Proteases Arm Coronavirus Spike Proteins». Activation of Viruses by Host Proteases, 16-02-2018, pàg. 71–98. DOI: 10.1007/978-3-319-75474-1_4. PMC: 7122371.
- ↑ 10,0 10,1 10,2 «S - Spike glycoprotein precursor - Severe acute respiratory syndrome coronavirus 2 (2019-nCoV) - S gene & protein» (en anglès). [Consulta: 6 novembre 2020].
- ↑ Li, Jiacheng; Ma, Xiaoliang; Guo, Shuai; Hou, Chengyu; Shi, Liping «A Hydrophobic-Interaction-Based Mechanism Triggers Docking between the SARS-CoV-2 Spike and Angiotensin-Converting Enzyme 2» (en anglès). Global Challenges, n/a, n/a, pàg. 2000067. DOI: 10.1002/gch2.202000067. ISSN: 2056-6646.
- ↑ 12,0 12,1 Tang, Tiffany; Bidon, Miya; Jaimes, Javier A.; Whittaker, Gary R.; Daniel, Susan «Coronavirus membrane fusion mechanism offers a potential target for antiviral development». Antiviral Research, 178, 2020-6, pàg. 104792. DOI: 10.1016/j.antiviral.2020.104792. ISSN: 0166-3542. PMC: 7194977. PMID: 32272173.
- ↑ 13,0 13,1 13,2 13,3 13,4 13,5 13,6 13,7 13,8 Salvatori, Giovanni; Luberto, Laura; Maffei, Mariano; Aurisicchio, Luigi; Roscilli, Giuseppe «SARS-CoV-2 SPIKE PROTEIN: an optimal immunological target for vaccines». Journal of Translational Medicine, 18, 03-06-2020. DOI: 10.1186/s12967-020-02392-y. ISSN: 1479-5876. PMC: 7268185. PMID: 32493510.
