SARS-CoV-2: diferència entre les revisions
#quarantena |
|||
| Línia 43: | Línia 43: | ||
Un estudi de [[metagenòmica]] publicat el 2019 indicava que el [[SARS-CoV]] (del qual el SARS-CoV-2 n'és una soca) era el coronavirus més abundant en una mostra de [[Pangolí malai|pangolins malais]].<ref>{{cite journal|vauthors=Liu P, Chen W, Chen JP|title=Viral Metagenomics Revealed Sendai Virus and Coronavirus Infection of Malayan Pangolins (Manis javanica)|journal=Viruses|volume=11|issue=11|pages=979|date=October 2019|pmid=31652964|pmc=6893680|doi=10.3390/v11110979}}</ref> El 7 de de febrer 2020 un grup d'investigaors de [[Guangzhou]] van descobrir una mostra de pangolí amb una seqüència d'àcid nucleic «99% idèntica» a la del SARS-CoV-2.<ref>{{cite journal|vauthors=Cyranoski D|title=Did pangolins spread the China coronavirus to people?|url=https://www.nature.com/articles/d41586-020-00364-2|journal=Nature|doi=10.1038/d41586-020-00364-2|date=7 February 2020}}</ref> No obstant això, al final es va aclarir que aquesta identitat només es referia a un domini d'unió al receptor de la proteïna S del virus i no a tot el genoma. Aquest fet contrasta amb els coronavirus que s'havien trobat abans en [[Civeta|civetes]], que sí compartien gran identitat amb el SARS-CoV, fet que portà a llur matança massiva després l'anterior [[epidèmia de SARS del 2002 al 2004]].<ref>{{cite journal|vauthors=Xiao K, Zhai J, Feng Y|title=Isolation and Characterization of 2019-nCoV-like Coronavirus from Malayan Pangolins|date=February 2020|journal=[[bioRxiv]]|type=preprint|doi=10.1101/2020.02.17.951335}}</ref><ref>{{Ref-publicació|article=Mystery deepens over animal source of coronavirus|url=https://www.nature.com/articles/d41586-020-00548-w|publicació=Nature|data=2020-02-26|pàgines=18–19|volum=579|doi=10.1038/d41586-020-00548-w|llengua=en|nom=David|cognom=Cyranoski}}</ref> Els pangolins estan protegits per la llei xinesa, però eren igualment capturats i distribuïts il·legalment per suplir l'encara existent demanda de [[medicina tradicional xinesa]].<ref>{{cite news|newspaper=The Telegraph|url=https://www.telegraph.co.uk/science/2016/03/15/pangolins-13-facts-about-the-worlds-most-hunted-animal/|last=Kelly|first=Guy|date=1 January 2015|title=Pangolins: 13 facts about the world's most hunted animal|access-date=9 March 2020}}</ref> |
Un estudi de [[metagenòmica]] publicat el 2019 indicava que el [[SARS-CoV]] (del qual el SARS-CoV-2 n'és una soca) era el coronavirus més abundant en una mostra de [[Pangolí malai|pangolins malais]].<ref>{{cite journal|vauthors=Liu P, Chen W, Chen JP|title=Viral Metagenomics Revealed Sendai Virus and Coronavirus Infection of Malayan Pangolins (Manis javanica)|journal=Viruses|volume=11|issue=11|pages=979|date=October 2019|pmid=31652964|pmc=6893680|doi=10.3390/v11110979}}</ref> El 7 de de febrer 2020 un grup d'investigaors de [[Guangzhou]] van descobrir una mostra de pangolí amb una seqüència d'àcid nucleic «99% idèntica» a la del SARS-CoV-2.<ref>{{cite journal|vauthors=Cyranoski D|title=Did pangolins spread the China coronavirus to people?|url=https://www.nature.com/articles/d41586-020-00364-2|journal=Nature|doi=10.1038/d41586-020-00364-2|date=7 February 2020}}</ref> No obstant això, al final es va aclarir que aquesta identitat només es referia a un domini d'unió al receptor de la proteïna S del virus i no a tot el genoma. Aquest fet contrasta amb els coronavirus que s'havien trobat abans en [[Civeta|civetes]], que sí compartien gran identitat amb el SARS-CoV, fet que portà a llur matança massiva després l'anterior [[epidèmia de SARS del 2002 al 2004]].<ref>{{cite journal|vauthors=Xiao K, Zhai J, Feng Y|title=Isolation and Characterization of 2019-nCoV-like Coronavirus from Malayan Pangolins|date=February 2020|journal=[[bioRxiv]]|type=preprint|doi=10.1101/2020.02.17.951335}}</ref><ref>{{Ref-publicació|article=Mystery deepens over animal source of coronavirus|url=https://www.nature.com/articles/d41586-020-00548-w|publicació=Nature|data=2020-02-26|pàgines=18–19|volum=579|doi=10.1038/d41586-020-00548-w|llengua=en|nom=David|cognom=Cyranoski}}</ref> Els pangolins estan protegits per la llei xinesa, però eren igualment capturats i distribuïts il·legalment per suplir l'encara existent demanda de [[medicina tradicional xinesa]].<ref>{{cite news|newspaper=The Telegraph|url=https://www.telegraph.co.uk/science/2016/03/15/pangolins-13-facts-about-the-worlds-most-hunted-animal/|last=Kelly|first=Guy|date=1 January 2015|title=Pangolins: 13 facts about the world's most hunted animal|access-date=9 March 2020}}</ref> |
||
=== Biologia estructural === |
|||
<br /> |
|||
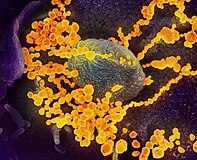
{{multiple image|align=right|direction=horizontal|total_width=400|image1=SARS-CoV-2 49531042877.jpg|alt1=SARS-CoV-2 emerging from a human cell|image2=SARS-CoV_scanning_electron_microscope_image.jpg|alt2=SARS-CoV-2 emerging from a human cell|footer_align=center|footer=[[Micrografia|Micrografies electròniques]] amb colors digitals del SARS-CoV-2 (groc) sorgides de cèl·lules humanes [[Cultiu cel·lular|cultivades]] en un laboratori.}} |
|||
[[Fitxer:Coronavirus_virion_structure.svg|miniatura|285x285px|Il·lustració d'un virió de SARS-CoV-2.]] |
|||
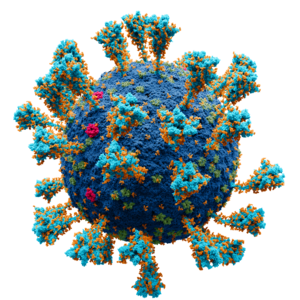
Cada [[virió]] del SARS-CoV-2 és aproximadament de 50–200 [[Nanometre|nanòmetres]] de diametre.<ref>{{cite journal|vauthors=Chen N, Zhou M, Dong X, Qu J, Gong F, Han Y, Qiu Y, Wang J, Liu Y, Wei Y, Sia J, You T, Zhang X, Zhang L|title=Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study|url=https://www.thelancet.com/journals/lancet/article/PIIS0140-6736(20)30211-7/fulltext|journal=[[The Lancet]]|volume=395|issue=10223|pages=507–513|display-authors=3|doi=10.1016/S0140-6736(20)30211-7|pmid=32007143|date=15 February 2020}}</ref> Com altres coronavirus, el SARS-CoV-2 té 4 proteïnes estructurals, conegudes com a proteïnes S (espiga), E (embolcall), M (membrana) i N ([[nucleocàpsida]]); la proteïna N manté el genoma de l’ARN, i les proteïnes S, E i M creen l’[[embolcall viral]].<ref name="WuStructure">{{cite journal|vauthors=Wu C, Liu Y, Yang Y, Zhang P, Zhong W, Wang Y, Wang Q, Xu Y, Li M, Li X, Zheng M, Chen L, Li H|display-authors=3|title=Analysis of therapeutic targets for SARS-CoV-2 and discovery of potential drugs by computational methods|journal=Acta Pharmaceutica Sinica B|date=Febrer del 2020|doi=10.1016/j.apsb.2020.02.008}}</ref> La proteïna de l'espiga, que s'ha fotografiat a nivell atòmic mitjançant [[Criomicroscopia electrònica|microscòpia electrònica criogènica]],<ref name="SCI-20200219">{{cite journal|vauthors=Wrapp D, Wang N, Corbett KS, Goldsmith JA, Hsieh CL, Abiona O, Graham BS, McLellan JS|display-authors=6|title=Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation|journal=Science|volume=367|issue=6483|pages=1260–1263|date=Febrer del 2020|pmid=32075877|doi=10.1126/science.abb2507}}</ref> és la proteïna responsable de permetre que el virus s’enganxi a la [[Membrana plasmàtica|membrana]] d’una cèl·lula hoste.<ref name="WuStructure" /> |
|||
Experiments de [[Predicció de l'estructura de les proteïnes|modelat de proteïnes]] sobre la proteïna espiga del virus aviat van suggerir que SARS-CoV-2 té una afinitat suficient amb els [[Enzim conversiu de l'angiotensina 2|receptors de l'enzim 2]] (ACE2) que converteixen l'angiotensina en cèl·lules humanes per utilitzar-los com a mecanisme d'[[Penetració (virus)|entrada de virions]].<ref>{{cite journal|vauthors=Xu X, Chen P, Wang J, Feng J, Zhou H, Li X, Zhong W, Hao P|display-authors=3|title=Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission|journal=Science China Life Sciences|volume=63|issue=3|pages=457–460|date=Març del 2020|pmid=32009228|doi=10.1007/s11427-020-1637-5}}</ref> El 22 de gener de 2020, un grup de la Xina que treballava amb el genoma del virus complet i un grup dels Estats Units que utilitzava mètodes de [[genètica inversa]] de manera independent i demostraven experimentalment que l'ACE2 podia actuar com a receptor del SARS-CoV-2.<ref>Zhou P, Yang XL, Wang XG, Hu B, Zhang L, Zhang W, et al. (Febrer del 2020). "A pneumonia outbreak associated with a new coronavirus of probable bat origin". ''Nature''. '''579''' (7798): 270–273. {{DOI|10.1038/s41586-020-2012-7}} {{PMID|32015507}}</ref><ref name="Letco22Jan2020">{{cite journal|vauthors=Letko M, Munster V|title=Functional assessment of cell entry and receptor usage for lineage B β-coronaviruses, including 2019-nCoV|date=Gener del 2020|journal=[[bioRxiv]]|type=preprint|doi=10.1101/2020.01.22.915660}}</ref> Els estudis han demostrat que el SARS-CoV-2 té una afinitat més alta per l'ACE2 humà que la soca del virus SARS original.<ref name="SCI-20200219" /> El SARS-CoV-2 també pot utilitzar [[basigina]] per obtenir entrada de cèl·lules.<ref>{{cite journal|vauthors=Wang K, Chen W, Zhou YS, Lian JQ, Zhang Z, Du P, Gong L, Zhang Y, Cui HY, Geng JJ, Wang B, Sun XX, Wang CF, Yang X, Lin P, Deng YQ, Wei D, Yang XM, Zhu YM, Zhang K, Zheng ZH, Miao JL, Guo T, Shi Y, Zhang J, Fu L, Wang QY, Bian H, Zhu P, Chen ZN|display-authors=3|title=SARS-CoV-2 invades host cells via a novel route: CD147-spike protein|journal=[[bioRxiv]]|type=preprint|doi=10.1101/2020.03.14.988345|year=2020}}</ref> |
|||
La preparació inicial de la proteïna espiga mitjançant proteasa transmembrana, la serina 2 ([[TMPRSS2]]) és essencial per a l’entrada de SARS-CoV-2.<ref name="HoffmanCell">{{cite journal|title=SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor|vauthors=Hoffman M, Kliene-Weber H, Krüger N, Herrler T, Erichsen S, Schiergens TS, Herrler G, Wu NH, Nitsche A, Müller MA, Drosten C, Pöhlmann S|display-authors=3|date=16 April 2020|journal=[[Cell (journal)|Cell]]|volume=181|pages=1–10|doi=10.1016/j.cell.2020.02.052|pmid=32142651}}</ref> Després que un virió de SARS-CoV-2 s’uneixi a una cèl·lula diana, la [[proteasa]] de la cèl·lula TMPRSS2 obre la proteïna de l'espiga del virus, exposant un [[pèptid de fusió]]. El virió després allibera ARN a la cèl·lula, obligant a la cèl·lula a produir còpies del virus que es difonen per infectar més cèl·lules.<ref name="econ-anatomy-killer">{{cite magazine|title=Anatomy of a Killer: Understanding SARS-CoV-2 and the drugs that might lessen its power|url=https://www.economist.com/briefing/2020/03/12/understanding-sars-cov-2-and-the-drugs-that-might-lessen-its-power|vauthors=|magazine=[[The Economist]]|date=12 de Març del 2020}}</ref><ref>{{cite journal|vauthors=Lu R, Zhao X, Li J, Niu P, Yang B, Wu H, Wang W, Song H, Huang B, Zhu N, Bi Y, Ma X, Zhan F, Wang L, Hu T, Zhou H, Hu Z, Zhou W, Zhao L, Chen J, Meng Y, Wang J, Lin Y, Yuan J, Xie Z, Ma J, Liu WJ, Wang D, Xu W, Holmes EC, Gao GF, Wu G, Chen W, Shi W, Tan W|display-authors=6|title=Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding|journal=[[The Lancet]]|volume=395|issue=10224|pages=565–574|date=February 2020|pmid=32007145|doi=10.1016/S0140-6736(20)30251-8}}</ref> El SARS-CoV-2 produeix almenys tres [[Factor de virulència|factors de virulència]] que afavoreixen el vessament de nous virions a partir de cèl·lules hostes i inhibeixen la [[resposta immune]].<ref name="WuStructure" /><ref name="Gralinski2020">{{cite journal|vauthors=Gralinski LE, Menachery VD|title=Return of the Coronavirus: 2019-nCoV|journal=Viruses|volume=12|issue=2|pages=135|date=January 2020|pmid=31991541|doi=10.3390/v12020135}}</ref> |
|||
== Referències == |
== Referències == |
||
{{Referències}} |
{{Referències}} |
||
Revisió del 19:43, 21 març 2020
|
|
Aquest article o secció tracta d'un fet d'actualitat. |
| Severe acute respiratory syndrome-related coronavirus | |
|---|---|
 | |
| Enregistrament | |
| Dades | |
| Hoste | |
| Genoma | virus d'ARN monocatenari positiu |
| Descobridor o inventor | Zhang Jixian |
| Malaltia | pandèmia de la COVID-19 i COVID-19 |
| Mida del genoma | 29.903 |
| Taxonomia | |
| Regne | Orthornavirae |
| Fílum | Pisuviricota |
| Classe | Pisoniviricetes |
| Ordre | Nidovirales |
| Família | Coronaviridae |
| Gènere | Betacoronavirus |
| Espècie | Severe acute respiratory syndrome-related coronavirus |
| Nomenclatura | |
| Epònim | Coronavirus relacionats amb la Síndrome respiratòria aguda greu |
 | |
 | |
| Tipus | Orthocoronavirinae |
|---|---|
| Epònim | Coronavirus relacionats amb la Síndrome respiratòria aguda greu |
| Patogènia | |
| Causa de | pandèmia de la COVID-19 i COVID-19 |
| Classificació | |
| CIM-11 | XN109 |
| Recursos externs | |
| MeSH | D000086402 |
| UMLS CUI | C5203676 |
El SARS-CoV-2, o coronavirus 2 del síndrome respiratori agut greu, inicialment anomenat 2019-nCov, és un tipus de coronavirus (gènere betacoronavirus) que va ser detectat per primer cop el desembre de 2019 i que és el causant de la COVID-19.[1][2][3] Té una similitud del 70% amb el virus de la SARS (SARS-CoV), del qual se'n considera una soca.[4] És contagiós entre humans i n'ha originat la pandèmia iniciada a la ciutat xinesa de Wuhan a finals del 2019.
El seu genoma està format per una única cadena ARN i se'l classifica com virus d'ARN monocatenari positiu. La primera seqüència genètica fou aïllada d'un pacient afectat per pneumònia a Wuhan i a partir d'aquí en seguiren moltes més.
Esdeveniments
El descobriment es va fer en tractar diverses persones de la ciutat de Wuhan, Xina afectades de pneumònia.[5] Les autoritats xineses van informar el 31 de desembre de 2019 sobre 27 casos.[6]
L'origen de la infecció es va localitzar inicialment en un mercat d'animals vius de la mateixa ciutat, que havia sigut el primer gran focus, però estudis posteriors van concloure que el virus havia de tenir altres orígens fora del mercat. El gener de 2020 les autoritats xineses van posar la ciutat de Wuhan en quarantena, prohibint als seus habitants sortir-ne o desplaçar-se. Dies posteriors es van aïllar fins a 10 ciutats, sumant un total de 33 milions de persones en quarantena.[7]
El gener de 2020 es van confirmar els primers casos fora de la Xina, detectant-se primer a Tailàndia i Japó i posteriorment se'n van detectar a Corea del Sud, Taiwan, Vietnam, Singapur, els EUA [6] i més properament a França.[8] La taxa de mortalitat s'ha establert en un 2.9% sobre 842 casos confirmats.
El dia 23 de gener es va reunir el Comitè d'Emergències del Reglament Sanitari Internacional de l'Organització Mundial de la Salut (OMS), que va concloure que, de moment, la malaltia no constituïa una emergència de salut pública mundial i va enviar una missió pluridisciplinària a treballar sobre el terreny.[9]
El dia 23 de febrer Itàlia prohibeix l'accés i la sortida d'11 municipis pel COVID-19, sumant fins a 50.000 persones confinades[10]. S'aixeca el confinament el 8 de març del 2020, però s'imposen en el mateix decret unes mesures més restrictives que afecten a 16 milions de persones i 14 províncies [11]
A data del 27 de febrer s'havien registrat 82.738 casos confirmats, dels quals, 78.514 casos dins de la Xina, amb 2.814 defuncions.[12]
El nombre d'infectats va anar creixent i el virus es va anar estenent per tots els països.
Símptomes i tractament

Els símptomes són febre, fatiga, tos seca i insuficiència respiratòria. Els casos més greus poden resultar en pneumònia, insuficiència renal i finalment, la mort.[13]
No hi ha cap tractament disponible i els tractaments es centren en alleujar els símptomes. Actualment s'està estudiant l'efectivitat d'antivirals existents, incloent-hi inhibidors de la proteasa com el indinavir, saquinavir, remdesivir, lopinavir / ritonavir i l'interferó beta.[14]
Virologia
Infecció
S'ha confirmat la transmissió d'humà a humà del virus, inclús durant el període d'incubació de la malaltia. Alguns grups han estimat un ritme de reproducció bàsic (R0) d'entre 3 i 5 (cada persona infectada pot infectar-ne de 3 a 5 de mitjana). Altres grups l'han estimat entre 1.4 i 3.8. S'ha comprovat que el virus es pot contagiar en cadenes de fins a 4 individus.[15]
Reservori
Els principals sospitosos de ser els reservoris del virus són animals vius venuts al mercat majorista de marisc de Huanan a Wuhan, ja que els primers humans infectats pel virus eren treballadors del mercat i, per tant, estaven fortament exposats al contacte amb diversos animals. En el brot de SARS del 2003 també es va identificar un mercat d'animals com el lloc d'inici de l'epidèmia.[16][17]
Arran de l'epidèmia de SARS del 2003, es van identificar i seqüenciar diversos coronavirus procedents de ratpenats, principalment de la família dels rinolòfids. El SARS-CoV-2 és d'aquest grup i dos coronavirus identificats el 2015 i 2017 presenten una similitud del 80% amb el nou virus.[18] També es va identificar un tercer coronavirus amb una semblança del 96%.[19]
Un estudi de metagenòmica publicat el 2019 indicava que el SARS-CoV (del qual el SARS-CoV-2 n'és una soca) era el coronavirus més abundant en una mostra de pangolins malais.[20] El 7 de de febrer 2020 un grup d'investigaors de Guangzhou van descobrir una mostra de pangolí amb una seqüència d'àcid nucleic «99% idèntica» a la del SARS-CoV-2.[21] No obstant això, al final es va aclarir que aquesta identitat només es referia a un domini d'unió al receptor de la proteïna S del virus i no a tot el genoma. Aquest fet contrasta amb els coronavirus que s'havien trobat abans en civetes, que sí compartien gran identitat amb el SARS-CoV, fet que portà a llur matança massiva després l'anterior epidèmia de SARS del 2002 al 2004.[22][23] Els pangolins estan protegits per la llei xinesa, però eren igualment capturats i distribuïts il·legalment per suplir l'encara existent demanda de medicina tradicional xinesa.[24]
Biologia estructural

Cada virió del SARS-CoV-2 és aproximadament de 50–200 nanòmetres de diametre.[25] Com altres coronavirus, el SARS-CoV-2 té 4 proteïnes estructurals, conegudes com a proteïnes S (espiga), E (embolcall), M (membrana) i N (nucleocàpsida); la proteïna N manté el genoma de l’ARN, i les proteïnes S, E i M creen l’embolcall viral.[26] La proteïna de l'espiga, que s'ha fotografiat a nivell atòmic mitjançant microscòpia electrònica criogènica,[27] és la proteïna responsable de permetre que el virus s’enganxi a la membrana d’una cèl·lula hoste.[26]
Experiments de modelat de proteïnes sobre la proteïna espiga del virus aviat van suggerir que SARS-CoV-2 té una afinitat suficient amb els receptors de l'enzim 2 (ACE2) que converteixen l'angiotensina en cèl·lules humanes per utilitzar-los com a mecanisme d'entrada de virions.[28] El 22 de gener de 2020, un grup de la Xina que treballava amb el genoma del virus complet i un grup dels Estats Units que utilitzava mètodes de genètica inversa de manera independent i demostraven experimentalment que l'ACE2 podia actuar com a receptor del SARS-CoV-2.[29][30] Els estudis han demostrat que el SARS-CoV-2 té una afinitat més alta per l'ACE2 humà que la soca del virus SARS original.[27] El SARS-CoV-2 també pot utilitzar basigina per obtenir entrada de cèl·lules.[31]
La preparació inicial de la proteïna espiga mitjançant proteasa transmembrana, la serina 2 (TMPRSS2) és essencial per a l’entrada de SARS-CoV-2.[32] Després que un virió de SARS-CoV-2 s’uneixi a una cèl·lula diana, la proteasa de la cèl·lula TMPRSS2 obre la proteïna de l'espiga del virus, exposant un pèptid de fusió. El virió després allibera ARN a la cèl·lula, obligant a la cèl·lula a produir còpies del virus que es difonen per infectar més cèl·lules.[33][34] El SARS-CoV-2 produeix almenys tres factors de virulència que afavoreixen el vessament de nous virions a partir de cèl·lules hostes i inhibeixen la resposta immune.[26][35]
Referències
- ↑ Press, Europa. «La OMS da nombre oficial al nuevo coronavirus, desde ahora será Covid-19», 11-02-2020. [Consulta: 23 febrer 2020].
- ↑ «COVID-19, nombre de la enfermedad del coronavirus» (en castellà).
- ↑ «Què saben els científics del nou coronavirus identificat a la Xina?». [Consulta: 24 gener 2020].
- ↑ Gorbalenya, Alexander E.; Baker, Susan C.; Baric, Ralph S.; de Groot, Raoul J.; Drosten, Christian «The species Severe acute respiratory syndrome-related coronavirus : classifying 2019-nCoV and naming it SARS-CoV-2» (en anglès). Nature Microbiology, 02-03-2020, pàg. 1–9. DOI: 10.1038/s41564-020-0695-z. ISSN: 2058-5276.
- ↑ S'inclouen els casos del vaixell Grand Princess.
- ↑ 6,0 6,1 «Ministerio de Sanidad, Consumo y Bienestar Social - Profesionales - Neumonía por un nuevo coronavirus (nCov) en China». [Consulta: 24 gener 2020].
- ↑ «La Xina restringeix els viatges a 10 ciutats i confina 33 milions de persones», 24-01-2020. [Consulta: 24 gener 2020].
- ↑ «Confirmats tres casos del coronavirus a França, els primers d'Europa», 24-01-2020. [Consulta: 26 gener 2020].
- ↑ El número mostra casos en el vaixell Diamond Princess, actualment en quarantena en aigües japoneses, però no estan inclosos en els recomptes del govern japonès.
- ↑ S'inclouen els casos del vaixell MS River Anuket.
- ↑ «Itàlia aïlla 16 milions de persones a la Llombardia i 14 províncies més», 08-03-2020. [Consulta: 8 març 2020].
- ↑ «Here's everything we know about the coronavirus outbreak so far» (en anglès americà), 21-02-2020. [Consulta: 23 febrer 2020].
- ↑ «Q&A on coronaviruses» (en anglès). Organització Mundial de la Salut. [Consulta: 27 gener 2020].
- ↑ Paules, Catharine I.; Marston, Hilary D.; Fauci, Anthony S. «Coronavirus Infections—More Than Just the Common Cold» (en anglès). JAMA, 23-01-2020. DOI: 10.1001/jama.2020.0757.
- ↑ «Statement on the meeting of the International Health Regulations (2005) Emergency Committee regarding the outbreak of novel coronavirus 2019 (n-CoV) on 23 January 2020» (en anglès). [Consulta: 27 gener 2020].
- ↑ Hui, David S.; Azhar, Esam I.; Madani, Tariq A.; Ntoumi, Francine; Kock, Richard «The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health — The latest 2019 novel coronavirus outbreak in Wuhan, China» (en anglès). International Journal of Infectious Diseases, 91, 01-02-2020, pàg. 264–266. DOI: 10.1016/j.ijid.2020.01.009. ISSN: 1201-9712. PMID: 31953166.
- ↑ Myers, Steven Lee «China’s Omnivorous Markets Are in the Eye of a Lethal Outbreak Once Again» (en anglès). The New York Times, 25-01-2020. ISSN: 0362-4331.
- ↑ «auspice». [Consulta: 27 gener 2020].
- ↑ Zhou, Peng; Yang, Xing-Lou; Wang, Xian-Guang; Hu, Ben; Zhang, Lei «Discovery of a novel coronavirus associated with the recent pneumonia outbreak in humans and its potential bat origin» (en anglès). bioRxiv, 23-01-2020, pàg. 2020.01.22.914952. DOI: 10.1101/2020.01.22.914952.
- ↑ «Viral Metagenomics Revealed Sendai Virus and Coronavirus Infection of Malayan Pangolins (Manis javanica)». Viruses, vol. 11, 11, October 2019, pàg. 979. DOI: 10.3390/v11110979. PMC: 6893680. PMID: 31652964.
- ↑ «Did pangolins spread the China coronavirus to people?». Nature, 07-02-2020. DOI: 10.1038/d41586-020-00364-2.
- ↑ «Isolation and Characterization of 2019-nCoV-like Coronavirus from Malayan Pangolins». bioRxiv, February 2020. DOI: 10.1101/2020.02.17.951335.
- ↑ Cyranoski, David «Mystery deepens over animal source of coronavirus» (en anglès). Nature, 579, 26-02-2020, pàg. 18–19. DOI: 10.1038/d41586-020-00548-w.
- ↑ Kelly, Guy «Pangolins: 13 facts about the world's most hunted animal». , 01-01-2015.
- ↑ «Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study». The Lancet, vol. 395, 10223, 15-02-2020, pàg. 507–513. DOI: 10.1016/S0140-6736(20)30211-7. PMID: 32007143.
- ↑ 26,0 26,1 26,2 «Analysis of therapeutic targets for SARS-CoV-2 and discovery of potential drugs by computational methods». Acta Pharmaceutica Sinica B, Febrer 2020. DOI: 10.1016/j.apsb.2020.02.008.
- ↑ 27,0 27,1 «Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation». Science, vol. 367, 6483, Febrer 2020, pàg. 1260–1263. DOI: 10.1126/science.abb2507. PMID: 32075877.
- ↑ «Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission». Science China Life Sciences, vol. 63, 3, Març 2020, pàg. 457–460. DOI: 10.1007/s11427-020-1637-5. PMID: 32009228.
- ↑ Zhou P, Yang XL, Wang XG, Hu B, Zhang L, Zhang W, et al. (Febrer del 2020). "A pneumonia outbreak associated with a new coronavirus of probable bat origin". Nature. 579 (7798): 270–273. doi:10.1038/s41586-020-2012-7 PMID: 32015507
- ↑ «Functional assessment of cell entry and receptor usage for lineage B β-coronaviruses, including 2019-nCoV». bioRxiv, Gener 2020. DOI: 10.1101/2020.01.22.915660.
- ↑ «SARS-CoV-2 invades host cells via a novel route: CD147-spike protein». bioRxiv, 2020. DOI: 10.1101/2020.03.14.988345.
- ↑ «SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor». Cell, vol. 181, 16-04-2020, pàg. 1–10. DOI: 10.1016/j.cell.2020.02.052. PMID: 32142651.
- ↑ «Anatomy of a Killer: Understanding SARS-CoV-2 and the drugs that might lessen its power». The Economist. 12 de Març del 2020.
- ↑ «Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding». The Lancet, vol. 395, 10224, February 2020, pàg. 565–574. DOI: 10.1016/S0140-6736(20)30251-8. PMID: 32007145.
- ↑ «Return of the Coronavirus: 2019-nCoV». Viruses, vol. 12, 2, January 2020, pàg. 135. DOI: 10.3390/v12020135. PMID: 31991541.
Enllaços externs
| A Wikimedia Commons hi ha contingut multimèdia relatiu a: SARS-CoV-2 |