Enzim: diferència entre les revisions
m Robot substituint el text: (-AND +ADN, -l•l +l·l, -’ +', -” +", -“ +", -‘ +', - I + i , -(I +(i , - I, + i,) |
|||
| Línia 125: | Línia 125: | ||
==Enllaços externs== |
==Enllaços externs== |
||
* [http://www.brenda-enzymes.org BRENDA] — comprehensive compilation of information and literature references about all known enzymes |
|||
{{Commonscat|Enzymes}} |
{{Commonscat|Enzymes}} |
||
Revisió del 10:50, 13 gen 2009
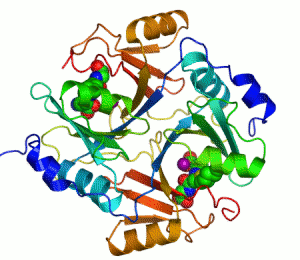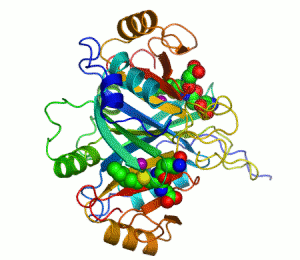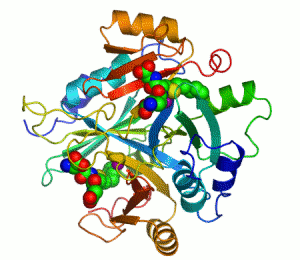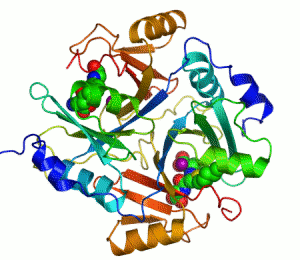
Els enzims són biomolècules que catalitzen (és a dir, augmenten la velocitat de) reaccions químiques.[1][2] Gairebé tots els enzims són proteïnes. En reaccions enzimàtiques, les molècules al principi del procés reben el nom de substrats, i l'enzim les converteix en molècules diferents, els productes. Gairebé tots els procesos de les cèl·lules necessiten la participació significativa d'enzims. Com que els enzims són extremament selectius quant al substrat i només catalitzen algunes reaccions d'entre moltes possibilitats, el conjunt d'enzims produïts en una cèl·lula determina les rutes metabòliques que hi tenen lloc.
Com tots els catalitzadors, els enzims funcionen reduint l'energia d'activació (Ea o ΔG‡) d'una reacció, augmentant així de manera dramàtica la velocitat de reacció. Com tots els catalitzadors, els enzims no són consumits per les reaccions que catalitzen, ni n'alteren l'equilibri químic. Tanmateix, els enzims difereixen d'altres catalitzadors en què són molt més específics. Se sap que els enzims catalitzen unes 4.000 reaccions bioquímiques.[3] Algunes molècules d'ARN anomenades ribozims també catalitzen reaccions, i en són un exemple important algunes parts del ribosoma.[4][5] Molècules sintètiques anomenades enzims artificials també presenten una catàlisi similar a la dels enzims.[6]
L'activitat dels enzims pot ser afectada per altres molècules. Els inhibidors són molècules que redueixen l'activitat enzimàtica; els activadors són molècules que l'augmenten. Molts medicaments i verins són inhibidors enzimàtics. L'activitat dels enzims també és afectada per la temperatura, el medi químic (per exemple, el pH), i la concentració del substrat. Alguns enzims tenen un ús comercial, per exemple, en la síntesi d'antibiòtics. A més, alguns productes de neteja utilitzen enzims per a catalitzar reaccions bioquímiques (per exemple, els enzims en detergents desfan les taques de proteïnes o greix de la roba; els enzims dels ablanidors de carn fragmenten les proteïnes, fent que la carn sigui més fàcil de mastegar).
Etimologia i història

Ja a finals del segle XVIII i principis del XIX, es coneixia la digestió de la carn per mitjà de secrecions estomacals[7] i la conversió de midó en sucres per mitjà d'extractes vegetals i saliva. Tanmateix, no s'havia identificat el mecanisme pel qual això passava.[8]
Al segle XIX, quan estudiava la fermentació de sucres en alcohol per mitjà de llevat, Louis Pasteur arribà a la conclusió que aquesta fermentació era catalitzada per una força vital continguda dins les cèl·lules del llevat, anomenada "ferment", que es creia que només funcionava en organismes vivents. Escrigué que "la fermentació de l'alcohol és un procés relacionat amb la vida i l'organització de les cèl·lules del llevat, i no amb la mort o putrefacció de les cèl·lules."[9]
El 1878, el fisiòleg alemany Wilhelm Kühne (1837-1900) fou el primer en utilitzar el mot "enzim", que prové del grec ἔνζυμον "en el llevat", per a descriure aquest procés. El mot "enzim" fou posteriorment utilitzat per a referir-se a substàncies no vivents com ara la pepsina, i el mot "ferment" per a referir-se a l'activitat química produïda pels organismes vivents.
El 1897 Eduard Buchner començà a estudiar la capacitat d'extractes de llevat que mancaven totalment de cèl·lules vivents de llevat per fermentar sucre. En una sèrie d'experiments a la Universitat de Berlín, descobrí que el sucre era fermentat fins i tot quan no hi havia cèl·lules vivents de llevat en la mescla.[10] Donà el nom de zimasa a l'enzim que havia causat la fermentació de la sucrose.[11] El 1907 rebé el Premi Nobel de Química "per la seva investigació en bioquímica i el seu descobriment de la fermentació acel·lular". Seguint l'exemple de Buchner, els enzims solen ser anomenats segons la reacció que duen a terme. Generalment s'afegeix el sufix -asa al nom dels substrats (per exemple, la lactasa és l'enzim que descompon la lactosa) o el tipus de reacció (per exemple, l'ADN polimerasa forma polímers d'ADN).
Havent quedat demostrat que els enzims podien funcionar a l'exterior d'una cèl·lula vivent, el pas següent era determinar-ne la naturalesa bioquímica. Molts dels primers investigadors remarcaren que l'activitat enzimàtica estava associada amb proteïnes, però alguns científics (com ara el premi Nobel Richard Willstätter) argumentaven que les proteïnes eren simplement portadores dels enzims autèntics i que per elles soles no eren capaces de dur a terme la catàlisi. Tanmateix, el 1926, James B. Sumner demostrà que l'enzim ureasa era proteïna pura i el cristal·litzà; Summer féu el mateix amb l'enzim catalasa el 1937. La conclusió que les proteïnes poden ser enzims queda demostrada definitivament per Northrop i Stanley, que treballaren en els enzims digestius pepsina (1930), tripsina i quimotripsina. Aquests tres científics foren guardonats amb el Premi Nobel de Química del 1946.[12]
El descobriment que els enzims podien cristal·litzar-se eventualment permeté determinar-ne l'estructura per mitjà de difraccio de rajos X. Això es féu per primer cop amb el lisozim, un enzim que es troba en les llàgrimes, la saliva i la clara dels ous que digereix les parets cel·lulars d'alguns bacteris; la seva estructura fou determinada per un grup encapçalat per David Chilton Phillips i publicada el 1965.[13] Aquesta determinació de l'estructura del lisozim a alta resolució marca el principi del camp de la biologia estructural i l'esforç per esbrinar com funcionen els enzims a nivell atòmic.
Estructures i mecanismes

Els enzims són generalment proteïnes globulars, i la seva mida va des de només 62 residus d'aminoàcids, en el cas del monòmer de 4-oxalocrotonat tautomerasa,[14] fins a més de 2.500 residus en l'àcid gras sintasa.[15] Existeix un nombre reduït de catalitzadors de base ARN, i el més comú és el ribosoma; reben el nom d'enzims ARN o ribozims. L'activitat dels enzims és determinada per la seva estructura tridimensional.[16] La majoria d'enzims són més grans que els substrats sobre els quals actuen, i només una petita part de l'enzim (aproximadament 3-4 aminoàcids) està directament implicada en la catàlisi.[17] La regió que conté aquests residus catalítics, s'adhereix al substrat, i després duu a terme la reacció és coneguda com a lloc actiu. Els enzims també poden contenir llocs que s'adhereixen a cofactors, que són necessaris per la catàlisi. Alguns enzims també contenen llocs d'adherència a petites molècules, que sovint són productes directes o indirectes, o substrats de la reacció catalitzada. Aquesta adherència pot servir per a augmentar o reduir l'activitat de l'enzim, cosa que possibilita una regulació realimentada.
Com totes les proteïnes, els enzims es formen com a llargues cadenes lineals d'aminoàcids que es pleguen per a produir un producte tridimensional. Cada seqüència d'aminoàcid produeix una estructura específica, amb les seves propietats particulars. En ocasions, poden agrupar-se cadenes proteíniques individuals per a formar un complex proteic. La majoria d'enzims poden ser desnaturalitzats – és a dir, desplegats i desactivats – per mitjà de desnaturalitzants calorífics o químics, que destrueixen l'estructura tridimensional de la proteïna. Segons l'enzim de què es tracti, la desnaturalització pot ser reversible o irreversible.
Especificitat
Els enzims són generalment molt específics quant a les reaccions que catalitzen i els substrats que estan implicats en aquestes reaccions. La forma complementària, la càrrega i les característiques hidròfiles/hidròfobes dels enzims i els substrats són les responsables d'aquesta especificitat. Els enzims també poden presentar nivells importants d'estereoespecificitat, regioselectivitat i quimioselectivitat.[18]
Alguns dels enzims més específics i precisos són els que estan implicats en la còpia i expressió del genoma. Aquests enzims tenen mecanismes de "control de qualitat". Un enzim com ara l'ADN polimerasa catalitza una reacció en una primera fase, i després verifica que el producte sigui correcte en una segona fase.[19] Aquest procés bifàsic resulta en una proporció mitjana d'error de menys d'un error per cada cent milions de reaccions, en el cas de polimerases mamiferoides d'alta fidelitat.[20] També hi ha mecanismes de control similars a l'ARN polimerasa,[21] les aminoacil ARNt sintetases[22] i els ribosomes.[23]
Alguns enzims productors de metabòlits secundaris són descrits com a promiscus, car poden actuar sobre una varietat relativament àmplia de substrats diferents. S'ha suggerit que aquesta especificitat de substrat àmplia és important per l'evolució de noves rutes biosintètiques.[24]
Model de "pany i clau"
Els enzims són molt específics, i Emil Fischer suggerí el 1894 que això era perquè tant l'enzim com el substrat tenen formes geomètriques específiques complementàries que encaixen exactament l'una amb l'altra.[25] Això és denominat sovint model de "pany i clau". Tanmateix, mentre que aquest model explica l'especificitat dels enzims, no explica l'estabilització de l'estat de transició que assoleixen els enzims. Ha estat demostrat que el model de "pany i clau" és incorrecte, i el model d'ajustament induït és la figura enzim-substrat-coenzim més acceptada actualment.
Model d'ajustament induït

El 1958, Daniel Koshland suggerí una modificació del model de "pany i clau"; com que els enzims són estructures bastant flexibles, el lloc actiu canvia de forma constantment degut a les interaccions amb el substrat, a mesura que el substrat interactua amb l'enzim.[26] Com a resultat, el substrat no s'uneix simplement a un lloc actiu rígid, sinó que les cadenes laterals d'aminoàcids que formen el lloc actiu són modelades en les posicions precises que permeten a l'enzim executar la seva funció catalítica. En alguns casos, com el de les glicosidases, la molècula del substrat també canvia lleugerament de forma quan entra al lloc actiu.[27] El lloc actiu continua canviant fins que el substrat està completament unit, moment en què queden determinades la forma i la càrrega finals.[28]
Mecanismes
Els enzims poden actuar de diferents formes, totes les quals redueixen la ΔG‡:[29]
- Reduint l'energia d'activació per mitjà de la creació d'un medi en què l'estat de transició és estabilitzat (ex., tensant la forma d'un substrat – unint la conformació de l'estat de transició de les molècules substrat/producte, l'enzim distorsiona els substrats unit en la seva forma d'estat de transició, reduint així la quantitat d'energia requerida per completar la transició).
- Reduint l'energia de l'estat de transició, però sense distorsionar el substrat, per mitjà de la creació d'un medi amb la distribució de càrrega contrària a la de l'estat de transició.
- Subministrant una ruta alternativa. Per exemple, reaccionant temporalment amb el substrat per formar un complex ES intermedi, cosa que seria impossible sense l'enzim.
- Reduint el canvi d'entropia de la reacció, agrupant els substrats en l'orientació correcta per la reacció. Considerar ΔH‡ per si sola ignora aquest efecte.
És interessant remarcar que aquest efecte entròpic implica una desestabilització de l'estat fonamental,[30] i la seva contribució a la catàlisi és relativament minsa.[31]
Estabilització de l'estat de transició
La comprensió de l'origen de la reducció de ΔG‡ exigeix que se sàpiga com estabilitzen els enzims el seu estat de transició més que l'estat de transició de la reacció no catalitzada. Aparentment, el mètode més eficient d'assolir una gran estabilització és l'ús d'efectes electrostàtics, en particular creant un medi polar relativament fix orientat vers la distribució de càrrega de l'estat de transició.[32] No existeix un tal ambient a la reacció no catalitzada en aigua.
Dinàmica i funció
Investigacions recents han millorat la comprensió de la connexió entre la dinàmica interna de l'enzim i el seu mecanisme de catàlisi.[33][34][35] La dinàmica interna d'un enzim és el moviment de les seves parts internes (ex., aminoàcids, un grup d'aminoàcids, una regió de bucle, una hèlix alfa, una capa beta veïna o fins i tot un domini sencer d'aquestes proteïnes). Aquests moviments es produeixen a diverses escales temporals que van de femtosegons a segons. Les xarxes de residus proteics dins l'estructura d'un enzim poden contribuir a la catàlisi per moviments dinàmics.[36][37][38][39] Els moviments proteics són vitals per molts enzims, però la importància relativa de les vibracions petites i ràpides, o els moviments conformacionals més grans i lents, depèn del tipus de reacció. Tanmateix, tot i que aquests moviments són importants en la unió i l'alliberament de substrats i productes, no està clar si els moviments proteics contribueixen a accelerar els passos químics a les reaccions enzimàtiques.[40] Aquests nous coneixements també tenen implicacions en la comprensió dels efectes al·lostèrics i el desenvolupament de nous medicaments.
Modulació al·lostèrica
Els enzims al·lostèrics canvien la seva estructura en resposta a la unió amb efectors. La modulació pot ser directa, quan l'efector s'uneix directament a llocs d'unió de l'enzim, o indirecta, quan l'efector s'uneix a altres proteïnes o subunitats de proteïnes que interactuen amb l'enzim al·lostèric i per tant influeixen l'activitat catalítica.
Cofactors i coenzims
Cofactors
Alguns enzims no requereixen cap component addicional per a executar plenament la seva activitat. En canvi, d'altres requereixen la unió amb molècules no proteiques anomenades cofactors per a actuar.[41] Els cofactors poden ser o bé compostos inorgànics (ex., ions de metalls o centres de ferro-sofre) o bé orgànics (ex., flavina i hemo). Els cofactors orgànics poden ser o bé grups prostètics, que són units fermament a un enzim, o coenzims, que són alliberats del lloc actiu de l'enzim durant la reacció. Els coenzims inclouen NADH, NADPH i el trifosfat d'adenosina. Aquestes molècules actuen transferint grups químics entre els enzims.[42]
Un exemple d'enzim que conté un cofactor és l'anhidrasa carbònica, que és mostrada al diagrama de llaços més amunt amb un cofactor de zinc unit com a part del seu lloc actiu.[43] Aquestes molècules fermament unides solen trobar-se al lloc actiu i estan implicades en la catàlisi. Per exemple, els cofactors flavina i hemo solen estar implicats en reaccions redox.
Els enzims que requereixen un cofactor però que no el tenen unit reben el nom d'apoenzims o apoproteïnes. Un apoenzim junt amb el seu o els seus cofactors rep el nom d'holoenzim (la forma activa). La majoria de cofactors no estan enllaçats de manera covalent a un enzim, però estan units molt fermament. Tanmateix, els grups prostètics orgànics poden ser units de manera covalent (ex., el tiamina pirofosfat en l'enzim piruvat deshidrogenasa). El terme holoenzim també es pot aplicar a enzims que contenen múltiples subunitats proteiques, com ara les ADN polimerases; en aquest cas, l'holoenzim és el complex complet que conté totes les subunitats necessàries per l'activitat.
Coenzims

Els coenzims són petites molècules orgàniques que transporten grups químics d'un enzim a un altre.[44] Algunes d'aquestes substàncies químiques, com ara la riboflavina, la tiamina i l'àcid fòlic, són vitamines, això és quan aquests compostos no poden fabricar-se al cos i han de ser adquirits de la dieta. Els grups químics transportats inclouen l'ió d'hidrur (H-) transportat per NAD o NADP+, el grup acetil transportat pel coenzim A, els grups formil, metenil o metil transportats per l'àcid fòlic i el grup metil transportat per l'S-adenosilmetionina.
Com que els coenzims canvien químicament com a conseqüència de l'acció enzimàtica, és útil considerar que els coenzims són una classe especial de substrats, o segons substrats, que són comuns a molts enzims diferents. Per exemple, es coneixen uns 700 enzims que utilitzen el coenzim NADH.[45]
Els coenzims solen ser regenerats, i les seves concentracions mantingudes a un nivell estable dins la cèl·lula; per exemple, NADPH és regenerat per la ruta del pentosa-fosfat i l'S-adenosilmetionina és regenerada per la metionina adenosiltransferasa.
Termodinàmica

Com tots els catalitzadors, els enzims no alteren la posició de l'equilibri químic de la reacció. Habitualment, en presència d'un enzim, la reacció transcorre en la mateixa direcció que ho faria sense l'enzim, simplement més ràpid. Tanmateix, en absència de l'enzim, altres possibles reaccions "espontànies" no catalitzades poden crear productes diferents, car en aquestes condicions aquest producte diferent és format més ràpidament.
A més, els enzims poden acoblar dues o més reaccions, de manera que una reacció termodinàmicament favorable pot ser utilitzada per a "alimentar" una de termodinàmicament desfavorable. Per exemple, la hidròlisi de l'ATP és sovint utilitzada per a alimentar altres reaccions químiques.
Els enzims catalitzen de manera igual les reaccions normals i les inverses. No alteren l'equilibri en si, només la velocitat a la qual s'assoleix. Per exemple, l'anhidrasa carbònica catalitza la seva reacció en qualsevol de les dues direccions segons la concentració dels seus reactants.
Tanmateix, si l'equilibri està molt desplaçat en una direcció, és a dir, en una reacció molt exergònica, la reacció és efectivament irreversible. En aquestes condicions, l'enzim només catalitzarà la reacció en la direcció permesa per la termodinàmica.
Cinètica

La cinètica enzimàtica és la investigació de com els enzims s'uneixen a substrats i els transformen en productes. Les dades de velocitat utilitzades en les anàlisis cinètiques són obtingudes d'assajos enzimàtics.
El 1902, Victor Henri[46] proposà una teoria quantitativa de la cinètica enzimàtica, però les seves dades experimentals no resultaren útils car la importància de la concentració d'ions d'hidrogen encara no era apreciada. Després que Peter Lauritz Sørensen definís l'escala logarítmica del pH i introduís el concepte d'emmagatzematge el 1909[47] el químic alemany Leonor Michaelis i la investigadora postdoctoral canadenca Maud Leonora Menten repetiren els experiments d'Henri i confirmaren la seva equació, que és denominada cinètica d'Henri-Michaelis-Menten (en ocasions també cinètica de Michaelis-Menten).[48] La seva obra fou continuada per George Edward Briggs i John Burdon Sanderson Haldane, que derivaren equacions cinètiques que encara són usades àmpliament avui en dia.[49]
La contribució principal d'Henri fou pensar en les reaccions enzimàtiques de dues etapes. A la primera, el substrat s'uneix reversiblement a l'enzim, formant el complex enzim-substrat. A vegades s'anomena això complex de Michaelis. Aleshores, l'enzim catalitza el pas químic de la reacció i allibera el producte.

Els enzims poden catalitzar fins a diversos milions de reaccions per segon. Per exemple, la reacció catalitzada per l'orotidina 5'-fosfat descarboxilasa consumeix la meitat del substrat en 78 milions d'anys si no hi ha cap enzim present. Tanmateix, quan s'afegeix la descarboxilasa, el mateix procés només triga 25 mil·lisegons.[50] La velocitat d'un enzim depèn de les condicions de la solució i de la concentració del substrat. Les condicions que desnaturalitzen la proteïna eliminen l'activitat enzimàtica, com ara temperatures elevades, un pH extrem o altes concentracions de sal, mentre que l'augmentació de la concentració del substrat tendeix a augmentar l'activitat. Per a determinar la velocitat màxima d'una reació enzimàtica, s'augmenta la concentració del substrat fins que s'observa una velocitat constant de formació del producte. La corba de saturació de la dreta mostra això. La saturació es produeix perquè, a mesura que augmenta la concentració del substrat, una part cada cop més important de l'enzim lliure és transformada en la forma ES unida al substrat. A la velocitat màxima (Vmax) de l'enzim, tots els llocs actius de l'enzim estan units al substrats, i la quantit de complex ES és la mateixa que la quantitat total d'enzim. Tanmateix, Vmax només és una de les constants cinètiques dels enzims. La quantitat de substrat necessària per a assolir una determinada velocitat de reacció també és important. Això és determinat per la constant de Michaelis-Menten (Km), que és la concentració del substrat necessària perquè un enzim assoleixi la meitat de la seva velocitat màxima. Cada enzimé una Km característica per un substrat donat, i això pot mostrar com de ferma és la unió del substrat a l'enzim. Una altra constant útil és kcat, que és el nombre de molècules de substrat que porta un lloc actiu per segon.
L'eficiència d'un enzim es pot expressar en termes de kcat/Km. Això també rep el nom de constant d'especificitat i incorpora la constant de velocitat de cada pas de la reacció. Com que la constant d'especificitat reflecteix tant l'afinitat com la capacitat catalítica, és útil per comparar enzims diferents entre ells, o el mateix enzim amb diferents substrats. El màxim teòric de la constant d'especificitat rep el nom de límit de difusió i és d'aproximadament 108 to 109 (M-1 s-1). En aquest punt, cada col·lisió de l'enzim amb el seu substrat resultarà en catàlisi, i la velocitat de formació del producte no està limitada per la velocitat de reacció sinó per la velocitat de difusió. Els enzims amb aquestes propietats són considerats "catalíticament perfectes" o "cinèticament perfectes". En són exemples la triosa-fosfat isomerasa, l'anhidrasa carbònica, l'acetilcolinesterasa, la catalasa, la fumarasa, la β-lactamasa, i la superòxid dismutasa.
La cinètica de Michaelis-Menten es basa en la llei de masses, que deriva de les assumpcions de difusió lliure i col·lisions aleatòries impulsades termodinàmicament. Tanmateix, molts processos bioquímics o cel·lulars es desvien significativament d'aquestes condicions, degut a apinyament molecular, separació en fases de l'enzim/substrat/producte, o moviment molecular mono o bidimensional.[51] En aquestes situacions, es pot aplicar una cinètica de Michaelis-Menten fractal.[52][53][54][55]
Alguns enzims funcionen amb una cinètica més ràpida que la velocitat de difusió, cosa que semblaria impossible. S'han suggerit diversos mecanismes per a explicar aquest fenomen. Es creu que algunes proteïnes acceleren la catàlisi agafant el seu substrat i preorientant-lo per mitjà de camps elèctrics dipolars. Altres models apel·len a una explicació basada en una tunelització mecanicoquàntica, segons la qual un protó o un electró pot travessar barreres d'activació, tot i que en el cas de la tunelització de protons el model roman bastant controvertit.[56][57] S'ha observat tunelització de protons en la triptamina.[58] Això suggereix que la catàlisi enzimàtica es podria descriure amb més precisió com a "a través de la barrera" en lloc del model tradicional, que requereix que el substrat passi "per sobre" d'una barrera energètica abaixada.
Inhibició


Referències
- ↑ Smith AD (Ed) i cols. (1997)) Oxford Dictionary of Biochemistry and Molecular Biology Oxford University Press. ISBN 0-19-854768-4
- ↑ Garrett RH, Grisham CM. (1999) Biochemistry, Second Edition Saunders College Publishing. 426-427. ISBN 0-03-022318-0
- ↑ Bairoch A. «The ENZYME database in 2000». Nucleic Acids Res, vol. 28, 2000, pàg. 304–305. DOI: 10.1093/nar/28.1.304. PMID: 10592255.
- ↑ Lilley D «Structure, folding and mechanisms of ribozymes». Curr Opin Struct Biol, vol. 15, 3, 2005, pàg. 313–23. DOI: 10.1016/j.sbi.2005.05.002. PMID: 15919196.
- ↑ Cech T «Structural biology. The ribosome is a ribozyme». Science, vol. 289, 5481, 2000, pàg. 878–9. DOI: 10.1126/science.289.5481.878. PMID: 10960319.
- ↑ Groves JT «Artificial enzymes. The importance of being selective». Nature, vol. 389, 6649, 1997, pàg. 329–30. DOI: 10.1038/38602. PMID: 9311771.
- ↑ de Réaumur, RAF «Observations sur la digestion des oiseaux». Histoire de l'academie royale des sciences, vol. 1752, 1752, pàg. 266, 461.
- ↑ Williams, H. S. (1904) A History of Science: in Five Volumes. Volume IV: Modern Development of the Chemical and Biological Sciences Harper and Brothers (New York) Consultat el 4 d'abril del 2007
- ↑ Dubos J. «Louis Pasteur: Free Lance of Science, Gollancz. Citat a Manchester K. L. (1995) Louis Pasteur (1822–1895)—chance and the prepared mind». Trends Biotechnol, vol. 13, 12, 1951, pàg. 511–515. DOI: 10.1016/S0167-7799(00)89014-9. PMID: 8595136.
- ↑ Biografia d'Eduard Buchner a http://nobelprize.org Consultat el 4 d'abril del 2007
- ↑ Text de la xerrada de Buchner a la cerimònia dels Premis Nobel del 1907 a http://nobelprize.org Consultat el 4 d'abril del 2007
- ↑ Guardonats del Premi Nobel de Química del 1946 a http://nobelprize.org Consultat el 4 d'abril del 2007
- ↑ Blake CC, Koenig DF, Mair GA, North AC, Phillips DC, Sarma VR. «Structure of hen egg-white lysozyme. A three-dimensional Fourier synthesis at 2 Angstrom resolution». Nature, vol. 22, 206, 1965, pàg. 757–761. DOI: 10.1038/206757a0. PMID: 5891407.
- ↑ Chen LH, Kenyon GL, Curtin F, Harayama S, Bembenek ME, Hajipour G, Whitman CP «4-Oxalocrotonate tautomerase, an enzyme composed of 62 amino acid residues per monomer». J. Biol. Chem., vol. 267, 25, 1992, pàg. 17716–21. PMID: 1339435.
- ↑ Smith S «The animal fatty acid synthase: one gene, one polypeptide, seven enzymes». Faseb J., vol. 8, 15, 1994, pàg. 1248–59. PMID: 8001737.
- ↑ Anfinsen C.B. «Principles that Govern the Folding of Protein Chains». Science, vol. 181, 1973, pàg. 223–230. DOI: 10.1126/science.181.4096.223. PMID: 4124164.
- ↑ The Catalytic Site Atlas a The European Bioinformatics Institute Consultat el 4 d'abril del 2007
- ↑ Jaeger KE, Eggert T. «Enantioselective biocatalysis optimized by directed evolution». Curr Opin Biotechnol., vol. 15(4), 2004, pàg. 305–313. DOI: 10.1016/j.copbio.2004.06.007. PMID: 15358000.
- ↑ Shevelev IV, Hubscher U. «The 3' 5' exonucleases». Nat Rev Mol Cell Biol., vol. 3, 5, 2002, pàg. 364–376. DOI: 10.1038/nrm804. PMID: 11988770.
- ↑ Berg J., Tymoczko J. and Stryer L. (2002) Biochemistry. W. H. Freeman and Company ISBN 0-7167-4955-6
- ↑ Zenkin N, Yuzenkova Y, Severinov K. «Transcript-assisted transcriptional proofreading». Science, vol. 313, 2006, pàg. 518–520. DOI: 10.1126/science.1127422. PMID: 16873663.
- ↑ Ibba M, Soll D. «Aminoacyl-tRNA synthesis». Annu Rev Biochem., vol. 69, 2000, pàg. 617–650. DOI: 10.1146/annurev.biochem.69.1.617. PMID: 10966471.
- ↑ Rodnina MV, Wintermeyer W. «Fidelity of aminoacyl-tRNA selection on the ribosome: kinetic and structural mechanisms». Annu Rev Biochem., vol. 70, 2001, pàg. 415–435. DOI: 10.1146/annurev.biochem.70.1.415. PMID: 11395413.
- ↑ Firn, Richard. «The Screening Hypothesis - a new explanation of secondary product diversity and function». [Consulta: 11 octubre 2006].
- ↑ Fischer E. «Einfluss der Configuration auf die Wirkung der Enzyme». Ber. Dt. Chem. Ges., vol. 27, 1894, pàg. 2985–2993. DOI: 10.1002/cber.18940270364.
- ↑ Koshland D. E. «Application of a Theory of Enzyme Specificity to Protein Synthesis». Proc. Natl. Acad. Sci., vol. 44, 2, 1958, pàg. 98–104. DOI: 10.1073/pnas.44.2.98. PMID: 16590179.
- ↑ Vasella A, Davies GJ, Bohm M. «Glycosidase mechanisms». Curr Opin Chem Biol., vol. 6, 5, 2002, pàg. 619–629. DOI: 10.1016/S1367-5931(02)00380-0. PMID: 12413546.
- ↑ Boyer, Rodney [2002]. «6». A: Concepts in Biochemistry. 2ª ed.. Nova York, Chichester, Weinheim, Brisbane, Singapur, Toronto.: John Wiley & Sons, Inc., 2002, p. 137–138. ISBN 0-470-00379-0. OCLC 51720783 [Consulta: 21 abril 2007].
- ↑ Fersht, A (1985) Enzyme Structure and Mechanism (2ª ed) p50–52 W H Freeman & co, Nova York ISBN 0-7167-1615-1
- ↑ Jencks W.P. "Catalysis in Chemistry and Enzymology." 1987, Dover, New York
- ↑ Villa J, Strajbl M, Glennon TM, Sham YY, Chu ZT, Warshel A «How important are entropic contributions to enzyme catalysis?». Proc. Natl. Acad. Sci. U.S.A., vol. 97, 22, 2000, pàg. 11899–904. DOI: 10.1073/pnas.97.22.11899. PMID: 11050223.
- ↑ Warshel A, Sharma PK, Kato M, Xiang Y, Liu H, Olsson MH «Electrostatic basis for enzyme catalysis». Chem. Rev., vol. 106, 8, 2006, pàg. 3210–35. DOI: 10.1021/cr0503106. PMID: 16895325.
- ↑ Eisenmesser EZ, Bosco DA, Akke M, Kern D. Enzyme dynamics during catalysis. Science. 2002 22 de febrer;295(5559):1520–3. PMID: 11859194
- ↑ Agarwal PK. Role of protein dynamics in reaction rate enhancement by enzymes. J Am Chem Soc. 2005 2 de novembre;127(43):15248-56. PMID: 16248667
- ↑ Eisenmesser EZ, Millet O, Labeikovsky W, Korzhnev DM, Wolf-Watz M, Bosco DA, Skalicky JJ, Kay LE, Kern D. Intrinsic dynamics of an enzyme underlies catalysis. Nature. 2005 3 de novembre;438(7064):117-21. PMID: 16267559
- ↑ Yang LW, Bahar I. «Coupling between catalytic site and collective dynamics: A requirement for mechanochemical activity of enzymes». Structure., vol. 13, Juny 2005, pàg. 893–904. DOI: 10.1016/j.str.2005.03.015. PMID: 15939021.
- ↑ Agarwal PK, Billeter SR, Rajagopalan PT, Benkovic SJ, Hammes-Schiffer S. «Network of coupled promoting motions in enzyme catalysis». Proc. Natl. Acad. Sci. U S A., vol. 99, Març 2002, pàg. 2794–9. DOI: 10.1073/pnas.052005999. PMID: 11867722.
- ↑ Agarwal PK, Geist A, Gorin A. Protein dynamics and enzymatic catalysis: investigating the peptidyl-prolyl cis-trans isomerization activity of cyclophilin A. Biochemistry. 2004 August 24;43(33):10605-18. PMID: 15311922
- ↑ Tousignant A, Pelletier JN. «Protein motions promote catalysis». Chem Biol., vol. 11, 8, Agost 2004, pàg. 1037–42. DOI: 10.1016/j.chembiol.2004.06.007. PMID: 15324804.
- ↑ Olsson M.H.M., Parson W.W. and Warshel A. "Dynamical Contributions to Enzyme Catalysis: Critical Tests of A Popular Hypothesis" Chem. Rev., 2006 105: 1737-1756
- ↑ de Bolster, M.W.G. «Glossary of Terms Used in Bioinorganic Chemistry». International Union of Pure and Applied Chemistry, 1997. [Consulta: 30 octubre 2007].
- ↑ de Bolster, M.W.G. «Glossary of Terms Used in Bioinorganic Chemistry». International Union of Pure and Applied Chemistry, 1997. [Consulta: 30 octubre 2007].
- ↑ Fisher Z, Hernandez Prada JA, Tu C, Duda D, Yoshioka C, An H, Govindasamy L, Silverman DN i McKenna R. «Structural and kinetic characterization of active-site histidine as a proton shuttle in catalysis by human carbonic anhydrase II». Biochemistry., vol. 44(4), 2005, pàg. 1097–115. DOI: 10.1021/bi0480279. PMID: 15667203.
- ↑ AF Wagner, KA Folkers (1975) Vitamins and coenzymes. Interscience Publishers New York| ISBN 0-88275-258-8
- ↑ BRENDA The Comprehensive Enzyme Information System Consultat el 4 d'abril del 2007
- ↑ Henri, V. «Theorie generale de l'action de quelques diastases». Compt. Rend. Hebd. Acad. Sci. Paris, vol. 135, 1902, pàg. 916–919.
- ↑ Sørensen,P.L. «Enzymstudien {II}. Über die Messung und Bedeutung der Wasserstoffionenkonzentration bei enzymatischen Prozessen». Biochem. Z., vol. 21, 1909, pàg. 131-304.
- ↑ Michaelis L., Menten M. «Die Kinetik der Invertinwirkung». Biochem. Z., vol. 49, 1913, pàg. 333–369. English translation Consultat el 6 d'abril del 2007
- ↑ Briggs G. E., Haldane J. B. S. «A note on the kinetics of enzyme action». Biochem. J., vol. 19, 1925, pàg. 339–339. PMID: 16743508.
- ↑ Radzicka A, Wolfenden R. «A proficient enzyme». Science, vol. 6, 267, 1995, pàg. 90–931. DOI: 10.1126/science.7809611. PMID: 7809611.
- ↑ Ellis RJ «Macromolecular crowding: obvious but underappreciated». Trends Biochem. Sci., vol. 26, 10, 2001, pàg. 597–604. DOI: 10.1016/S0968-0004(01)01938-7. PMID: 11590012.
- ↑ Kopelman R «Fractal Reaction Kinetics». Science, vol. 241, 4873, 1988, pàg. 1620–26. DOI: 10.1126/science.241.4873.1620.
- ↑ Savageau MA «Michaelis-Menten mechanism reconsidered: implications of fractal kinetics». J. Theor. Biol., vol. 176, 1, 1995, pàg. 115–24. DOI: 10.1006/jtbi.1995.0181. PMID: 7475096.
- ↑ Schnell S, Turner TE «Reaction kinetics in intracellular environments with macromolecular crowding: simulations and rate laws». Prog. Biophys. Mol. Biol., vol. 85, 2–3, 2004, pàg. 235–60. DOI: 10.1016/j.pbiomolbio.2004.01.012. PMID: 15142746.
- ↑ Xu F, Ding H «A new kinetic model for heterogeneous (or spatially confined) enzymatic catalysis: Contributions from the fractal and jamming (overcrowding) effects». Appl. Catal. A: Gen., vol. 317, 1, 2007, pàg. 70–81. DOI: 10.1016/j.apcata.2006.10.014.
- ↑ Garcia-Viloca M., Gao J., Karplus M., Truhlar D. G. «How enzymes work: analysis by modern rate theory and computer simulations». Science, vol. 303, 5655, 2004, pàg. 186–195. DOI: 10.1126/science.1088172. PMID: 14716003.
- ↑ Olsson M. H., Siegbahn P. E., Warshel A. «Simulations of the large kinetic isotope effect and the temperature dependence of the hydrogen atom transfer in lipoxygenase». J. Am. Chem. Soc., vol. 126, 9, 2004, pàg. 2820–1828. DOI: 10.1021/ja037233l. PMID: 14995199.
- ↑ Masgrau L., Roujeinikova A., Johannissen L. O., Hothi P., Basran J., Ranaghan K. E., Mulholland A. J., Sutcliffe M. J., Scrutton N. S., Leys D. «Atomic Description of an Enzyme Reaction Dominated by Proton Tunneling». Science, vol. 312, 5771, 2006, pàg. 237–241. DOI: 10.1126/science.1126002. PMID: 16614214.
- ↑ Cleland, W.W. «The Kinetics of Enzyme-catalyzed Reactions with two or more Substrates or Products 2. {I}nhibition: Nomenclature and Theory». Biochim. Biophys. Acta, vol. 67, 1963, pàg. 173-187.
Vegeu també
Enllaços externs
- BRENDA — comprehensive compilation of information and literature references about all known enzymes
Plantilla:Enllaç AD Plantilla:Enllaç AD Plantilla:Enllaç AD Plantilla:Enllaç AD Plantilla:Enllaç AD Plantilla:Enllaç AD Plantilla:Enllaç AD


